-
微卫星又称为简单序列重复(SSR), 短串联重复(STR), 是以1~个碱基为重复单位组成的短串联重复DNA序列[1]。微卫星分子标记是基于PCR技术发展起来的一种分子遗传标记技术, 具有重复性好、共显性遗传、多态性位点含量高、实验操作简单、检测方法简便、对DNA质量要求不高等优点[2]。在亲本分析, 基因图谱构建, 确立遗传和进化关系, 遗传多样性分析等研究中得到了广泛的应用[3]。微卫星依据其来源分为两类: 基因组微卫星(GenomicSSR)和表达序列标签微卫星(SSR, EST-SSR) [4]。EST-SSR是基于EST序列开发的SSR, 相对于Genomic SSR引物开发过程简单, 成本低, 在物种间通用性较强[5]。随着基因组的发展, EST序列的数目在公共数据库内迅速增长(http://www.ncbi.nlm.Nih.gov), 为SSR标记提供了一个庞大的资源[2, 6]。
沙棘属(Hippophae)属于胡颓子科(Elaeagnaceae), 是多年生落叶灌木、小乔木或乔木, 风媒传粉, 雌雄异株, 具有固氮作用, 适应性极强, 主要分布于中欧和中亚地区[7]。依据Sun等[7]和Bartish等[8]学者的系统分类, 分为7个种和8个亚种。沙棘是集生态效益、经济效益和社会效益为一体的多用途树种, 因其果实富含Vc和沙棘油, 枝叶含高蛋白, 根系有根瘤等特点备受瞩目[9]。近些年, 随着DNA分子标记技术的不断发展与完善, 针对沙棘遗传多样性、亲缘关系和品种鉴定等的研究得到进展[10], 如利用RFLP、ITS、cpDNA等分子标记对沙棘进行了研究[7-8-11], 但利用SSR分子标记对沙棘的研究较少, 同时目前开发可用的沙棘SSR引物较少, 仅有孙燕琳等[12]利用葡萄SSR引物筛选适用于沙棘的SSR引物, Wang等[13]利用磁珠富集法开发的9对SSR引物和Jain等[14]利用EST开发的11对EST-SSR引物。为了丰富沙棘的SSR分子标记, 本研究通过对向阳沙棘转录组序列进行分析, 开发出大量EST-SSR标记, 为沙棘亲本分析、遗传多样性、遗传育种等研究提供支持。
-
本研究中涉及转录组测序的沙棘品种“向阳”(H.rhamnoides subsp. mongolica al Xiangyang)是俄罗斯专家通过天然蒙古沙棘选育所得。随机选取采自新疆青河县莫齐克村的天然种群蒙古沙棘中的16个个体, 采用植物全基因组DNA提取试剂盒(北京天根生物公司)从其叶片中提取总DNA, 用于引物筛选和多样性检测。
-
沙棘EST序列来源于本实验室对“向阳”品种的转录组测序所得。收集向阳沙棘新鲜根、茎、叶提取RNA, 构建cDNA文库, 再利用Illumina Hiseq2000测序平台(IlluminaInc)进行高通量测序, 最后用Trinity[15]软件将测序序列拼接成无冗余的独立基因集, 即由EST序列组成的一个转录组。该实验由北京诺禾致源生物信息科技有限公司完成。所得转录组序列已提交到NCBI数据库, 序列号为SRP067785。
-
利用软件MISA(http://pgrc.ipk-gatersleben.de/misa/misa.html)检测转录组序列中简单重复序列, 确定SSR位点。SSR的搜索标准为: 单、二、三、四、五和六核苷酸的最少重复次数分别为10、6、5、5、5、5, 同时搜索复合SSR。对不同SSR类型在转录组中的密度分布进行统计。
根据SSR位点两端的保守区域, 利用Primer 3[16]设计引物用于SSR位点扩增。具体参数如下: 引物长度为20~28 bp, 最适长度为24 bp; (G+C)含量为40%~65%, 最适为50%。随机挑选179对预期扩增产物为100~300 bp、重复单元为二或者三核苷酸的SSR引物, 交由上海生工生物工程技术服务有限公司合成。
-
以8个蒙古沙棘个体的DNA为模板, 用上述179对引物进行PCR扩增筛选。PCR反应体系为10 μL: 去离子水3.6 μL, 2×MasterMix(艾德莱)5 μL; 10 μmol·L-1的F和R端引物各0.2 μL, 模板DNA1 μL (约50~100 ng)。PCR反应在ABI System 9700 (Applied Biosystems, Life Technologies)上进行, 具体程序如下: 94 ℃预变性4 min, 30个循环的94 ℃变性30 s, 55 ℃复性30 s, 72 ℃延伸45 s, 最后72 ℃延伸10 min。利用1.2%的琼脂糖凝胶对PCR产物进行检测。
筛选出能够扩增出目标片段的引物后, 利用16个蒙古沙棘个体进行多态性检测。为节约成本, 采用通用M13引物标记荧光的方法[17]进行PCR扩增和检测。PCR反应体系20 μL: 去离子水13.5 μL, 10×PCR缓冲液2 μL, 2.5 mnol·L dNTP 1.6 μL, 10 μmol·L-1的M13荧光引物0.8 μL, 10 μmol·L-1的F和R端引物分别为0.8 μL和0.2 μL, 5 U·μL-1 LrTaq DNA聚合酶(大连宝生物)0.1 μL, 模板DNA1 μL (约50~100 ng)。PCR反应在ABI System 9700PCR扩增仪(Applied Biosystems, Life Technologies)上进行, 反应条件为: 94 ℃预变性4 min, 30个循环的94 ℃变性30 s, 56 ℃复性30 s, 72 ℃延伸45 s, 另加8个循环的94 ℃变性30 s, 53 ℃复性30 s, 72 ℃延伸45 s, 最后72 ℃延伸10 min。将4种带有不同荧光标记(Fam, Hex, Rox和Tamra, 上海生工)的PCR产物各0.3 μL与9.0 μL的甲酰胺, 0.5 μL的Liz500混合后94℃变性5 min, 变性产物在ABI 3130测序仪上进行毛细管电泳, 最后利用GeneMapper version 4.0 (Applied Biosystems, Life Technologies)分析电泳结果得到基因型数据。
-
从筛选出具有多态性(等位基因数≥2)的引物中, 进一步筛选在蒙古沙棘个体中扩增成功率高、且上机检测峰图明晰的EST-SSR位点。利用GENALEX 6软件计算每个位点在这16个沙棘个体中的等位基因数量、观测杂合度(Ho)和期望杂合度(HE)。
-
对沙棘转录组中的EST序列去冗余后共收集17 383条具有SSR位点的序列, 其中包括复合SSR类型序列828条, 因其特殊性不计入SSR类型分析。在非复合的SSR类型中, 单核苷酸重复类型数量最多, 所占比例达到62.77%(10 392);其次是二核苷酸和三核苷酸重复类型, 所占比例分别为21.82%(3 613)和13.77%(2 279)。四核苷酸、五核苷酸、六核苷酸重复类型较少, 分别仅占1.45%(240)、0.09%(15)、0.10%(16)。
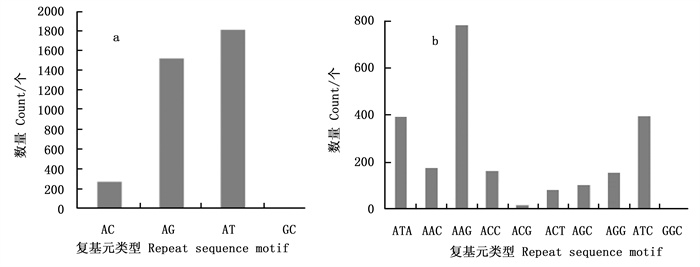
在所检测到的3 613个二核苷酸重复EST-SSR中, AT和AG两种类型的重复基元出现的次数较多, 分别为1 814个(50.21%)和1 521(42.10%)(图 1a)。在检测到的2 279个三核苷酸重复中, AAG为优势类型, 共783个(34.36%), ATC和ATA两种类型次之, 分别为400个(17.55%)和395个(17.33%)(图 1b)。
-
利用Primer 3.0为9 291条EST序列设计了适宜的SSR扩增引物, 剩余的8 092条序列没能得到合适的引物。从设计的引物中随机挑选179对二核苷酸、三核苷酸重复的EST-SSR引物进行PCR扩增检测, 发现142个位点能够在预期片段大小的位置扩增出清晰的条带, 扩增效率为79.33%。
选择92个位点的引物进一步结合M13荧光引物扩增16个蒙古沙棘个体, 并在ABI 3 130测序仪上检测各位点扩增产物(图 3), 发现40个位点在这些个体的扩增产物中找到2个以上的等位基因(表 1), 即呈现出多态, 多态位点比率为43.48%。从这40个多态位点中, 挑选出17个扩增效果稳定、且峰图清晰的EST-SSR位点, 其在所分析的天然蒙古沙棘种群中的等位基因个数为2~6个不等, 观测杂合度(HO)为0.083~0.875, 期望杂合度(HE)在0.180~0.750;分析得到该蒙古沙棘种群各位点平均期望观测杂合度为0.488, 平均期望杂合度为0.514。这17个位点的引物序列信息、扩增结果及多态性等见表 1。
表 1 沙棘17个多态EST-SSR位点的引物信息
Table 1. Information of 17 Hippophae EST-SSR loci in transcriptome sequences and tested with 16 sample
引物编号
Primer pairs引物序列(5'-3')
Primer sequence (5' -3')重复基元
Repeat motif退火温度
Annealing temperatures/℃等位基因数量
Number of alleles等位基因大小
Allele size range/bp观测杂合度
(HO)Observed heterozygosity期望杂合度
(HE) Expected heterozygosityHs008 F: ACTCATGCCCATCACCTTTT (AT) 6 55 3 238 ~242 0.467 0.580 R: GCTCGTTGCCACTGTTACAA Hs032 F: GCAGTCCGGACAGTCAGAAA (GTT)7 55 2 268 ~274 0.800 0.498 R: CCAAAGCAAAACCACGCAGA Hs047 F: GTGCAAAAACCAGGAGTGCC (TAG)6 55 2 184 ~188 0.600 0.491 R: TACAACCCTGCTACCCCCAT HS097 F: CAGCCGCCCTCTGACTAAAA (AT)6 55 4 280 ~294 0.083 0.733 R: GCTTCGAGGAAGAATTACAGGGA Hsl00 F: CTTGGCTGCCCATCTCGATT (TA) 8 55 6 259 ~274 0.875 0.672 R: ACGAATAAACCCCAGAACAGT Hsll1 F: ACAACTCAGTCCGTACATATGGT (AG) 9 55 4 163 ~177 0.818 0.649 R: GCGGCTGGAATCATCTTGGA Hsl46 F: ACAAGCGTGTGAGGATTCTT (CTA)5 55 2 142 ~146 0.500 0.492 R: CGATGAGAGGCCGGCATATT Hsl49 F: ACATTCCCCTTTCACAGACCC (TAT)6 55 2 138 ~142 0.636 0.483 R:CCAGTTGGGATTTTCTTGTTTGAGT Hs177 F: TGCGTCTTGATTTGGCACTTC (AC) 8 55 2 271 ~273 0.286 0.500 R: ACTCCAGGGTTCTCAAAGGA Hs192 F: TCGCTTCAAAAGGGAACAACA (AT)7 55 4 251 ~265 0.500 0.469 R: GCACACATAACACCCGCTTC Hsl95 F: TCCTCCTGACTGTCTCGACC (TA)7 55 2 247 ~249 0.200 0.180 R: CCCTAGTGAAGCTCAGTTTTGC Hs227 F: CCTTAGCATCACCAAAGCGC (ACA)6 55 2 160 ~168 0.273 0.236 R: CCTTGTGTGCACATTTCTGCA Hs278 F: CGCAGCAGGTTGACAGACTA (CT) 9 55 4 170 ~176 0.500 0.451 R: TCAAACAACGCACTTTGGGC Hs282 F: TGTCTTGCTACTCCTCCTGC (CA) 8 55 3 269 ~273 0.556 0.593 R: ACAGCGTGTTAGGAAAGTTGC Hs284 F: CCCATCTTGCCTCATGCTGT (AT) 8 55 5 269 ~285 0.667 0.750 R: ACCACAAGTCACGGAAATCCT Hs296 F: TGAACGCTCATGATCAAAACGT (TGA)5 55 3 225 ~234 0.400 0.515 R: CAGCTGGTGGAAGTTGAGGT Hs297 F: TCATGCTCACTTCCGTTGCT (CAC)6 55 2 156 ~162 0.133 0.444 R: AGGATTGAGCGGCGTGTTAA 平均Mean 3 0.488 0.514 -
本研究通过对具有SSR位点的17 383条EST序列的分析发现, 在沙棘转录组序列中, 单核苷酸重复类型的SSR最多, 其次是二核苷酸重复类型和三核苷酸重复; 四核苷酸, 五核苷酸和六核苷酸重复类型的总和不到1.7%。由于单核苷酸的特殊性, 大多数研究不将其作为SSR的研究对象[18]。因此, 忽略单核苷酸重复不计, 沙棘的EST-SSR位点以二核苷酸和三核苷酸重复类型为主, 这与目前大多数植物的研究一致[19]。先前研究表明, 二核苷酸重复在许多种的基因组中是常见类型, 但是在编码区出现的频率低于非编码区, 编码区常见的重复类型为三核苷酸。这可能是因为编码区的序列为密码子序列, 随着三核苷酸的增加或减少会改变阅读框架[6], 但是不会引起移码突变[20], 而本研究中, 二核苷酸重复类型所占比例高于三核苷酸重复类型, 不同于胡杨EST-SSR的研究[20]。这可能是由于不同研究搜索SSR的标准(SSR重复类型、次数、长度等)不同造成的差异[19]。
在沙棘二核苷酸重复基元的EST-SSR位点中, AT和AG是出现次数最多的两种类型, 分别占50.21%和42.10%;在三核苷酸重复基元中, AAG为优势重复类型, 占34.36%, ATC与ATA基元所占比例之和将近35%。沙棘的重复基元的主要类型与白桦、银杏、杨树、北美鹅掌楸、火炬树、蒙古栎等木本植物基本相同[2], 与水稻、高粱、玉米等作物存在差异, 不同物种之间重复基元存在的差异可能与各研究所用EST来源及数目不同, 也可能与物种转录组的特异性等有关[21]。
-
本研究设计的EST-SSR引物在天然蒙古沙棘种群中扩增效率较高, 达到79.33%。剩余引物无法扩增的原因可能是: 基因组DNA存在许多内含子片段, SSR位点所设计的一端或两端引物的位置刚好位于内含子或者外显子剪切点, 致使引物在基因组中找不到结合位点[19]。另外, 在引物的初筛过程中统一采用55 ℃退火进行PCR, 这也可能造成最适退火温度低的引物无法成功扩增。
判断SSR分子标记可用性的主要依据是其多态性, 本研究通过对天然蒙古沙棘种群的16个个体分析, 发现40对具有多态性的引物, 多态比率为43.48%, 低于胡杨(P. euphratica Oliv., 52.5%)[20]和中华猕猴桃(Actinidia chinensis Planch., 81.25%)[22], 高于银杏(Ginkgo biloba Linn., 31.9%)[23]。对17个扩增稳定且上机峰图明细的位点进一步分析, 得到来自新疆的天然蒙古沙棘种群遗传多样性处于中等水平(HO =0.488, HE=0.514)。由于沙棘为雌雄异株, 所以沙棘种群的遗传多样性相对较高, 不同位点多样性水平差别较大, 但是这些位点的平均多样性水平仍高于Wang等[13]利用磁珠法开发的基因组SSR所估算的中国沙棘亚种的多样性。这说明利用编码区的SSR位点估计的遗传多样性水平不一定低于基因组SSR位点。
-
本研究利用EST序列成功开发了40对具有多态性的沙棘引物。利用蒙古沙棘的16个样品对2种开发的引物进行多态性水平的检验和多态性相关分析, 发现利用序列数据开发的引物多样性高且多态性信息含量相对比较丰富, 可为沙棘属植物进行遗传多样性分析、遗传图谱构建系统发育分析和分子育种等多个方面的研究提供有力的支持
基于沙棘转录组序列开发EST-SSR分子标记
Development of EST-SSR Markers Based on Seabuckthorn Transcriptomic Sequences
-
摘要:
目的 本研究通过对蒙古沙棘“向阳”品种的转录组序列进行SSR引物开发,为沙棘亲本分析、遗传多样性和遗传育种等研究提供支持。 方法 利用已测得的转录组序列进行分析和筛选,整理具有SSR位点的序列,进行引物设计,随机挑选179条引物进行验证。 结果 得到具有SSR位点的EST序列17 383条,其中,单核苷酸、二核苷酸和三核苷酸重复基元分别占62.77%、21.82%和13.77%;二核苷酸重复基元类型以AT和AG为主,三核苷酸重复基元类型以AAG、ATC和ATA为主。9 291条序列设计出扩增EST-SSR位点的引物,随机合成的179对引物中,142对扩增成功,选取其中92个位点的扩增产物上机检测,40个SSR位点(43.48%)呈现多态。对扩增稳定且峰图清晰的17个多态位点的进一步分析, 得到蒙古沙棘天然群体在各位点的观测杂合度(HO)为0.083~0.875,期望杂合度(HE)为0.180~0.750。 结论 本研究开发出的EST-SSR标记,为后期进行沙棘属植物遗传多样性分析、遗传图谱构建及分子育种等研究提供了重要支持。 Abstract:Objective The transcriptomes ofHippophae rhamnoides subsp. mongolica cv'Xiangyang' were analyzed to design primers and develop EST-SSR (Expressed Sequence Tags-Simple Sequence Repeat) markers. Method The primers were designedand the SSR was developed based on the Expressed Sequence. 179 pairs of primers were validated randomly. Result 17 383 SSR-ESTs (SSR-containing EST) were identified. Of the total EST-SSRs, the mononucleotiderepeat was the most dominant type, accounting for 62.77%, followed by dinucleotiderepeats and trinucleotiderepeats, accounting for 21.82% and 13.77%, respectively. AT and AG were the most abundant dinucleotide motifs; AAG, ATC and ATA were repeated dominant motifs in trinucleotide. Based on these SSR-ESTs, 9 291 pairs of EST-SSR primers were designed; 179 pairs of primers were randomly selected and compounded for PCR amplification, among which 142 loci were amplified successfully. Amplificationproductions of 92 loci were genotyped using the DNA analyzer, of which 40 loci (43.48%) were identified as polymorphism. At last, 17 loci that amplified highly successfully and genotyped easily were recommend and analyzed for natural H. rhamnoides subsp. mongolica individuals, with observedheterozygosity (HO) ranged from 0.083 to 0.875 and expected heterozygosity (HE) ranged from 0.180 to 0.750. Conclusion These EST-SSR markers would be valuable for further research on genetic diversity analysis, linkage mapping construction and molecular breeding of the genus Hippophae. -
Key words:
- Hippophae rhamnoides
- / transcriptome
- / EST-SSR
- / primer
-
表 1 沙棘17个多态EST-SSR位点的引物信息
Table 1. Information of 17 Hippophae EST-SSR loci in transcriptome sequences and tested with 16 sample
引物编号
Primer pairs引物序列(5'-3')
Primer sequence (5' -3')重复基元
Repeat motif退火温度
Annealing temperatures/℃等位基因数量
Number of alleles等位基因大小
Allele size range/bp观测杂合度
(HO)Observed heterozygosity期望杂合度
(HE) Expected heterozygosityHs008 F: ACTCATGCCCATCACCTTTT (AT) 6 55 3 238 ~242 0.467 0.580 R: GCTCGTTGCCACTGTTACAA Hs032 F: GCAGTCCGGACAGTCAGAAA (GTT)7 55 2 268 ~274 0.800 0.498 R: CCAAAGCAAAACCACGCAGA Hs047 F: GTGCAAAAACCAGGAGTGCC (TAG)6 55 2 184 ~188 0.600 0.491 R: TACAACCCTGCTACCCCCAT HS097 F: CAGCCGCCCTCTGACTAAAA (AT)6 55 4 280 ~294 0.083 0.733 R: GCTTCGAGGAAGAATTACAGGGA Hsl00 F: CTTGGCTGCCCATCTCGATT (TA) 8 55 6 259 ~274 0.875 0.672 R: ACGAATAAACCCCAGAACAGT Hsll1 F: ACAACTCAGTCCGTACATATGGT (AG) 9 55 4 163 ~177 0.818 0.649 R: GCGGCTGGAATCATCTTGGA Hsl46 F: ACAAGCGTGTGAGGATTCTT (CTA)5 55 2 142 ~146 0.500 0.492 R: CGATGAGAGGCCGGCATATT Hsl49 F: ACATTCCCCTTTCACAGACCC (TAT)6 55 2 138 ~142 0.636 0.483 R:CCAGTTGGGATTTTCTTGTTTGAGT Hs177 F: TGCGTCTTGATTTGGCACTTC (AC) 8 55 2 271 ~273 0.286 0.500 R: ACTCCAGGGTTCTCAAAGGA Hs192 F: TCGCTTCAAAAGGGAACAACA (AT)7 55 4 251 ~265 0.500 0.469 R: GCACACATAACACCCGCTTC Hsl95 F: TCCTCCTGACTGTCTCGACC (TA)7 55 2 247 ~249 0.200 0.180 R: CCCTAGTGAAGCTCAGTTTTGC Hs227 F: CCTTAGCATCACCAAAGCGC (ACA)6 55 2 160 ~168 0.273 0.236 R: CCTTGTGTGCACATTTCTGCA Hs278 F: CGCAGCAGGTTGACAGACTA (CT) 9 55 4 170 ~176 0.500 0.451 R: TCAAACAACGCACTTTGGGC Hs282 F: TGTCTTGCTACTCCTCCTGC (CA) 8 55 3 269 ~273 0.556 0.593 R: ACAGCGTGTTAGGAAAGTTGC Hs284 F: CCCATCTTGCCTCATGCTGT (AT) 8 55 5 269 ~285 0.667 0.750 R: ACCACAAGTCACGGAAATCCT Hs296 F: TGAACGCTCATGATCAAAACGT (TGA)5 55 3 225 ~234 0.400 0.515 R: CAGCTGGTGGAAGTTGAGGT Hs297 F: TCATGCTCACTTCCGTTGCT (CAC)6 55 2 156 ~162 0.133 0.444 R: AGGATTGAGCGGCGTGTTAA 平均Mean 3 0.488 0.514 -
[1] Tautz D. Hypervariabflity of simple sequences as a general source for polymorphic DNA markers[J]. Nucleic acids research, 1989, 17(16): 6463-6471. doi: 10.1093/nar/17.16.6463 [2] 许玉兰, 蔡年辉, 康向阳, 等. EST-SSR标记的开发及其在木本植物中的分布特点[J]. 中国农学通报, 2012, 28(4): 1-7. [3] Kalia R K, Rai M K, Kalia S, et al. Microsatellite markers: an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3): 309-334. doi: 10.1007/s10681-010-0286-9 [4] 程小毛, 黄晓霞. SSR标记开发及其在植物中的应用[J]. 中国农学通报, 2011, 27(5): 304-307. [5] 赵罕, 朱高浦, 刘梦培, 等. 微卫星分子标记及其在林业中的应用[J]. 世界林业研究, 2013, 26(6): 21-26. [6] Ueno S, Moriguchi Y, Uchiyama K, et al. A second generation framework for the analysis of microsatellites in expressed sequence tags and the development of EST-SSR markers for a conifer[J]. Cryptomeria japonica. BMC Genomics, 2012, 13: 136. doi: 10.1186/1471-2164-13-136 [7] Sun K, Chen X, Ma R, et al. Molecular phylogenetics of Hippophae L. (Elaeagnaceae)based on the internal transcribed spacer(ITS)sequences of nrDNA[J]. Plant Systematics and Evolution, 2002, 235(1): 121-134. doi: 10.1007/s00606-002-0206-0 [8] Bartish I V, Jeppsson N, Nybom H, et al. Phylogeny of Hippophae(Elaeagnaceae)inferred from parsimony analysis of chloroplast DNA and morphology[J]. Systematic Botany, 2002, 27(1): 41-54. [9] 齐虹凌, 于泽源, 李兴国. 沙棘研究概述[J]. 沙棘, 2005, 18(2): 37-41. [10] 李蓉, 于永涛. DNA分子标记及在沙棘遗传育种中的应用展望[J]. 国际沙棘研究与开发, 2008, 6(2): 19-25. doi: 10.3969/j.issn.1672-4836.2008.02.007 [11] Wang A, Schluetz F, Liu J. Molecular evidence for double maternal origins of the diploid hybrid Hippophae goniocarpa(Elaeagnaceae)[J]. Botanical Journal of the Linnean Society, 2008, 156(1): 111-118. doi: 10.1111/j.1095-8339.2007.00729.x [12] 孙燕琳, 阮成江, 金华. 沙棘SSR分子标记的开发[J]. 安徽农业科学, 2007, 35(1): 45-46. doi: 10.3969/j.issn.0517-6611.2007.01.023 [13] Wang A, Zhang Q, Wan D, et al. Nine microsatellite DNA primers for Hippophae rhamnoides ssp. sinensis(Elaeagnaceae)[J]. Conservation Genetics, 2008, 9(4): 969-971. doi: 10.1007/s10592-007-9416-x [14] Jain A, Ghangal R, Grover A, et al. Development of EST-based new SSR markers in seabuckthorn[J]. Physiology and Molecular Biology of Plants, 2010, 16(4): 375-378. doi: 10.1007/s12298-010-0037-3 [15] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature biotechnology, 2011, 29(7): 644-652. doi: 10.1038/nbt.1883 [16] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature biotechnology, 2011, 29(7): 644-652. doi: 10.1038/nbt.1883 [17] Schuelke M. An economic method for the fluorescent labeling of PCR fragments[J]. Nature biotechnology, 2000, 18(2): 233-234. doi: 10.1038/72708 [18] 贯春雨, 张含国, 张磊, 等. 基于松科树种EST序列的落叶松SSR引物开发[J]. 东北林业大学学报, 2011, 39(1): 20-23. doi: 10.3969/j.issn.1000-5382.2011.01.007 [19] 高丽霞. 桑树EST—SSR引物开发[J]. 南方农业学报, 2013, 44(8): 1254-1257. doi: 10.3969/j:issn.2095-1191.2013.8.1254 [20] Du F K, Xu F, Qu H, et al. Exploiting the transcriptome of Euphrates Poplar, Populus euphratica(Salicaceae)to develop and characterize new EST-SSR markers and construct an EST-SSR database[J]. PloS one, 2013, 8(4): e61337. doi: 10.1371/journal.pone.0061337 [21] 李文娟, 马宏, 李正红, 等. 基于芭蕉属EST序列的地涌金莲SSR引物开发[J]. 林业科学研究, 2012, 25(2): 111-116. doi: 10.3969/j.issn.1001-1498.2012.02.001 [22] 孟蒙, 唐维, 刘嘉, 等. 基于中华猕猴桃"红阳" 转录组序列开发EST-SSR分子标记[J]. 应用与环境生物学报, 2014, 20(4): 564-570. [23] 李广平, 曹福亮. 银杏EST-SSR引物开发[J]. 西北农业学报, 2012, 21(12): 149-152. doi: 10.7606/j.issn.1004-1389.2012.12.029 -




 下载:
下载: