-
在林木遗传育种研究中, 分子标记是使用最广泛的一种遗传标记类型, 因其不受发育时期及环境或季节性的制约, 被广泛地被运用于遗传多样性、分子遗传育种等研究中[1-2]。SRAP(Sequence-related amplified polymorphism)即相关序列扩增多态性是一种新型分子标记技术, 其基础来源于PCR扩增反应, 通过对基因的开放阅读框(ORFs)中相对保守的外显子区域及变异丰富的内含子及启动子区域进行扩增, 获得物种或个体之间的遗传差异与分化[3-4]。该分子标记采用的是双引物设计, 因其简便、高效、产率高等特点, 使该标记成功地应用于遗传多样性分析、遗传图谱的构建等分子研究方面的分析[5-8]。
红椿(Toona ciliata Roem), 又名红楝子, 是楝科(Meliaceae)香椿属(Toona)速生植物。该树种材质优良, 纹理优美, 由于其木材呈现红色, 坚韧耐腐, 常作为高档家具用材出口国际, 开发潜力巨大, 被誉为“中国桃花心木”[9-10]。由于环境的变化、天然更新能力较差、过度的人为破坏及需求量的不断增加, 红椿数量在持续减少, 已成为珍贵的濒危树种, 被列为国家Ⅱ级重点保护野生植物。红椿在印度、老挝、缅甸、泰国、巴基斯坦至澳大利亚东海岸也有天然分布[11], 并在世界多地进行引种[12]。在我国集中分布在华南、华中、华东及西南地区, 呈零星或散点分布[13]。一个物种生存或者适应环境, 获得发展和进化的依靠前提是其自身所具有的遗传多样性[14]。研究红椿的遗传多样性对其选择育种、遗传资源的保护具有重要的理论和实践意义。
本研究采用单因素试验及正交试验对SRAP-PCR体系中的5大因素, 包括模板DNA、引物、Taq DNA聚合酶、Mg2+及dNTP进行优化, 旨在确定最优的稳定反应体系, 并从随机引物组合中筛选出能够扩增稳定、清晰且含有多态性条带的最优引物组合, 为进一步开展红椿遗传多样性研究、种质资源鉴定及遗传育种奠定基础, 提供技术支持。
-
以不同种源红椿母树叶片作为试材, 于采集地收集并利用硅胶迅速干燥, 置于实验室-80℃冰箱用于后续DNA提取。用于引物筛选的种源包括云南永仁、贵州望谟、四川会东、广东乐昌、江西宜丰、安徽黄山、福建南平及湖南城步。种源信息见表 1。
表 1 红椿SRAP-PCR体系优化试验材料
Table 1. Experimental material of T. ciliata for optimizing SRAP-PCR system
编号
No.种源代码
provenance省份
province纬度
latitude/N经度
longitude/E海拔
altitudeP1 乐昌 广东 25°07' 113°20' 359 P2 黄山 安徽 30°16' 118°08' 499 P3 望谟 贵州 25°10' 106°05' 500 P4 永仁 云南 25°01' 101°32' 1 539 P5 宜丰 江西 27°19' 115°26' 337 P6 城步 湖南 27°14' 111°28' 737 P7 南平 福建 26°38' 118°10' 800 P8 会东 四川 27°23' 102°09' 1 862 -
取100 mg干燥叶片做样品, 首先利用CTAB free溶液(0.2 mmol·L-1 Tris-HCl, 0.25 mmol·L-1 NaCl, 0.05 mmol·L-1 EDTA, pH=8.0)对样品进行低温洗涤, 去除杂质[15-16]。后采用EZNA® High-Performance DNA Mini Ki试剂盒提取红椿基因组DNA。使用0.8%琼脂糖凝胶电泳及超微量紫外分光光度计(Thermo NanoDrop 1000)进行检测。稀释至50 ng·μL-1, 样品置于-20℃下保存备用。
-
首先对反应体系的5大因素, 即, 模板DNA、引物、Taq DNA聚合酶、Mg2+及dNTP进行单因素试验。每个影响因素分别设置8个梯度。模板DNA浓度包括0、25、50、60、70、80、90和100 ng; 引物浓度包括0、0.1、0.2、0.3、0.4、0.5、0.6和0.7 μmol·L-1; Taq DNA聚合酶包括0、0.5、0.75、1、1.25、1.5、1.75和2 U; Mg2+浓度包括0、1、1.5、2、2.5、3、3.5和4 mmol·L-1; dNTP浓度梯度包括0、0.1、0.15、0.2、0.25、0.3、0.35和0.4 mmol·L-1。
-
进行单因素试验确定5大因素浓度范围后, 在其范围内选定4个水平浓度梯度(表 2), 采用L16(45)正交试验设计(表 3), 共16个处理, 每个处理选用不同种源DNA进行2次重复。根据所扩增出的电泳条带的多少、清晰度及背景颜色, 对16个处理进行评分[17-18]。最优的得16分, 最差的得1分, 两次重复取平均分为该处理水平扩增结果的最终得分。后计算每个因素在不同水平下的平均得分, 进行统计分析后获得极差。在某水平下某因素所参与的扩增反应所得到的扩增结果评分的总和用K值表示; R表示为极差, 即, 某试验因素在不同处理水平下所得到评分的最大平均值与最小平均值之差[19]。红椿SRAP分子标记反应总体系选用为25 μL(均含有2.5 μL 10×Buffer, 用重蒸水补齐体系), 引物选用M22(TGA GTA CAA ACC GG GTC)E27(GAC TGC GTA CGA ATT CCA)。
表 2 正交试验各因子水平设计
Table 2. The design of factors and the levels for orthogonal experimental
因子
factor水平level 1 2 3 4 Taq酶/U 1 1.25 1.5 1.75 Mg2+/(mmol·L-1) 1.5 2 2.5 3 dNTP/(mmol·L-1) 0.15 0.2 0.25 0.3 引物Primer/(μmol·L-1) 0.2 0.3 0.4 0.5 DNA模板/ng 25 50 75 100 表 3 SRAP-PCR正交试验设计[L16(45)]
Table 3. L16(45)orthogonal experimental of SRAP-PCR
处理编号 Taq DNA酶/(U) Mg2+/(mmol·L-1) dNTP/(mmol·L-1) 引物/(μmol·L-1) DNA模板/ng 1 1 1.5 0.15 0.2 25 2 1 2 0.2 0.3 50 3 1 2.5 0.25 0.4 75 4 1 3 0.3 0.5 100 5 1.25 1.5 0.2 0.4 100 6 1.25 2 0.15 0.5 75 7 1.25 2.5 0.3 0.2 50 8 1.25 3 0.25 0.3 25 9 1.5 1.5 0.25 0.5 50 10 1.5 2 0.3 0.4 25 11 1.5 2.5 0.15 0.3 100 12 1.5 3 0.2 0.2 75 13 1.75 1.5 0.3 0.3 75 14 1.75 2 0.25 0.2 100 15 1.75 2.5 0.2 0.5 25 16 1.75 3 0.15 0.4 50 -
SRAP分子标记反应程序选用为94℃ 5 min; 5个循环(94℃ 1 min, 35℃ 1 min, 72℃ 1 min); 35个循环(94℃ 1 min, 50℃ 1 min, 72℃ 1 min); 72℃延伸10 min; 4℃保存。产物在2%的琼脂糖凝胶上进行分离, 自动凝胶成像仪上照相保存, 用于体系优化因素确定。
-
在确定最优稳定体系后, 根据SRAP分子标记引物组合通用性的特点, 查找其在各物种研究中所发表的引物序列[20-26], 包括43个正向引物与35个反向引物, 随机组合, 共计1 505对引物组合对8份来源不同, 地理距离差别较大的红椿基因组DNA进行引物筛选。PCR扩增产物在6%变性聚丙烯酰胺凝胶上进行分离并通过银染染色检测。保鲜膜封胶, 照相或制干胶保存。
采用遗传多样性信息含量(PIC)评价试验所选定引物组合评估能力。
$ \mathrm{PIC}_{i}=2 f_{i}\left(1-f_{i}\right) $
式中, PICi为标记i的遗传多样性信息含量, fi为标记i出现条带的频率, 1-fi是缺失条带的频率, 一对引物的PIC为各个条带的平均值[27]。
-
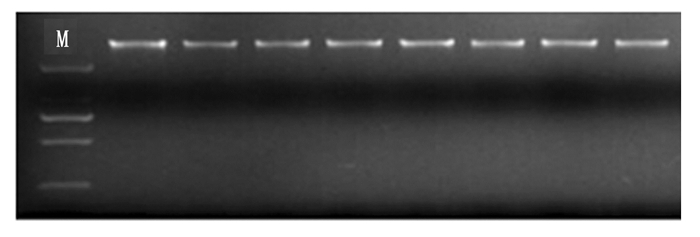
经超微量紫外分光光度计检测可知, 所提取的DNA OD260/OD280比值在1.7~1.9之间, OD260/OD230比值在1.8~3.0之间, 说明DNA纯度较高。电泳结果表明(图 1), 所提取的DNA均呈现一条清晰完整的主带, 无拖尾和降解, 无明显的RNA存在, 并且DNA浓度较高, 完整性好, 可用于下游SRAP分析。
-
因素浓度的优化主要目的是为了得到更加清晰、完整的条带, 并且要尽可能地将多态性条带扩增出来。红椿SRAP-PCR单因素试验结果表明, 5大因素的浓度范围较大, 25 μL体系中, 模板DNA浓度在25~100 ng范围内均可以扩增出清晰条带; 0.2~0.5 μmol·L-1引物浓度, 0.5~2 U Taq DNA聚合酶, 1~3 mmol·L-1 Mg2+ 浓度及0.15~0.3 mmol·L-1 dNTP浓度梯度均可扩增出多态性较大, 清晰完整、效果较好的条带, 所以将以上各因素的浓度作为正交试验的浓度范围进行设计。
-
在单因素试验所确定的5大因素的浓度范围内, 选定4个水平的浓度梯度, 根据L16(45)正交设计对SRAP-PCR进行体系优化。根据电泳结果首先进行直接主观性观察(图 3)。按照扩增条带的质量, 包括扩增清晰度、条带亮度及条带数目等对各组合进行评分, 两次重复取平均分(表 4)。

图 3 SRAP反应体系正交试验结果(A与B为两个重复)
Figure 3. Amplified pattern of SRAP-PCR based on orthogonal designs(A and B for two repeated trials)
表 4 不同处理得分情况表
Table 4. The result of range analysis for the factors
处理treatment 重复1 copy 1 重复2 copy 2 均值mean 1 6 7 6.5 2 15 14 14.5 3 13 13 13 4 14 15 14.5 5 1 2 1.5 6 2 1 1.5 7 9 9 9 8 12 12 12 9 3 3 3 10 11 11 11 11 16 16 16 12 10 10 10 13 7 6 6.5 14 5 5 5 15 8 8 8 16 4 4 4 最优条件及各因素影响力的大小由极差(R值)的大小判断。R值越大, 证明某一因素在不同水平下对反应体系及扩增结果影响越显著。结果显示(表 5), 在确定的4个水平内, 模板DNA、引物、Taq DNA聚合酶、Mg2+ 及dNTP 5大因素对扩增影响从大到小依次排列为: Mg2+ > Taq DNA聚合酶 > 引物 > dNTP > 模板DNA浓度。
表 5 各因素极差分析结果
Table 5. The result of range analysis for the factors
水平
levelTaq DNA聚合酶 Mg2+ dNTP 引物primer 模板DNA浓度
DNA concentration总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 1 48.5 12.125 17.5 4.375 28 7 30.5 7.625 37.5 9.375 2 24 6 32 8 34 8.5 49 12.25 30.5 7.625 3 40 10 46 11.5 33 8.25 29.5 7.375 31 7.75 4 23.5 5.875 40.5 10.125 41 10.25 27 6.75 37 9.25 极差range 25 6.25 28.5 7.125 13 3.25 22 5.5 7 1.75 各因素的某一水平影响反应体系的情况需要由K值确定, K值越大, 因素所处当前水平PCR扩增效果越好。根据表 4, 选定各因素K值最大值时的浓度作为最优体系: 其中模板DNA浓度为25 ng, 引物浓度为0.3 μmol·L-1、Taq DNA聚合酶为1 U、Mg2+ 浓度为2.5 mmol·L-1及dNTP浓度为0.3 mmol·L-1。
-
利用以上确定的最优体系进行重复试验, 确定体系的稳定性和适用性。利用8个不同种源的红椿基因组DNA进行检测。如图 4所示, 该体系扩增条带清晰可见, 并且多态性高。证明所得到的优化SRAP-PCR体系稳定并且可靠, 可有效地进行PCR扩增, 适用性强, 用于区别不同种源红椿之间的遗传差异, 可用于后续试验。

图 4 优化的SRAP-PCR体系对8份红椿DNA扩增结果
Figure 4. Amplification results of 8 different T. ciliata DNA with the best SRAP-PCR
根据条带扩增的多态性及清晰度进行引物组合的确定。最终筛选出30对引物组合(表 6), 用于红椿遗传多样性测定及分析。30对引物对红椿30个种源进行测定评估, 各引物组合所扩增出的条带、多态性条带、多态位点百分率和每对引物的PIC见表。根据表中多态百分率和PIC值进行判定, 引物组合Me35Em32(多态百分率为94.74%), Me23Em22, Me27Em7, Me31Em24, Me32Em17, Me39Em32和Me43Em19(以上引物组合的PIC值(0.43, 排名均为30对引物组合的前三名)可作为红椿SRAP分子标记的优先骨干引物。
表 6 30对SRAP引物的总条带数、多态性条带数、多态性比率及PIC值
Table 6. Total number of bands, polymorphic bands, polymorphism and polymorphic information content (PIC) obtained from 30 SRAP primer combinations
引物组合primer 总条带数 多态性条带 多态百分率/% PIC值 Me13Em8 22 15 68.18 0.37 Me14Em6 23 17 73.91 0.40 Me17Em21 27 21 77.78 0.38 Me19Em13 25 21 84.00 0.42 Me19Em15 26 23 88.46 0.42 Me19Em18 17 11 64.71 0.41 Me19Em27 25 21 84.00 0.41 Me22Em21 26 21 80.77 0.41 Me22Em27 28 20 71.43 0.41 Me23Em22 30 26 86.67 0.44 Me23Em31 28 21 75.00 0.41 Me27Em7 25 21 84.00 0.44 Me27Em18 40 31 77.5 0.39 Me27Em28 25 21 84.00 0.39 Me28Em18 36 27 75.00 0.40 Me31Em24 36 29 80.56 0.43 Me32Em17 28 20 71.43 0.43 Me32Em26 32 28 87.50 0.42 Me33Em27 23 11 47.83 0.35 Me34Em13 25 19 76.00 0.39 Me35Em13 27 15 55.56 0.42 Me35Em14 26 15 57.69 0.40 Me35Em32 27 14 51.85 0.45 Me38Em21 24 19 79.17 0.39 Me38Em24 29 21 72.41 0.42 Me38Em32 28 26 92.88 0.42 Me39Em32 19 18 94.74 0.43 Me40Em13 28 23 82.14 0.42 Me40Em22 21 15 71.43 0.33 Me43Em19 33 26 78.79 0.43 合计Total 809 616 平均值 Mean 26.97 20.53 75.85 0.41 相比较琼脂糖凝胶的分离效果, 6%变性聚丙烯酰胺凝胶分离效果更好, 银染染色检测条带清晰, 分辨率高, 更适合红椿SRAP分子标记的分析和鉴定。
-
本研究中所用试验材料均为多年生母树叶片, 全部取自红椿天然林分, 为野外获得。在运输过程中易受到DNA酶及酚类的影响, 导致DNA的降解, 而DNA的质量直接影响到SRAP-PCR反应。为避免或降低DNA降解情况发生, 本研究利用硅胶进行叶片迅速干燥后尽快置于-80℃低温冰箱保存, 减缓了叶片的氧化。红椿叶片中含有大量的酚类、糖类及次生代谢产物[28], 首先使用CTAB free溶液进行低温洗涤, 可达到去除杂质的目的, 后利用试剂盒提取法对DNA进行提取, 纯化了DNA的质量。经检测, 其OD260/OD280比值在1.7~1.9之间, 证明并无RNA的污染; OD260/OD230比值在1.8~3.0之间, 说明多糖及酚类物质得到有效去除, 所得到的DNA纯度较高, 在SRAP后续试验中可以有效的进行扩增, 提高扩增效率。
SRAP分子标记主要针对基因开放阅读框(ORFs)的特定区域进行扩增。上游引物长17 bp, 主要目标为基因中相对保守的外显子区域; 下游引物长18 bp, 富含AT-片段, 对多态性较为丰富的内含子和启动子进行扩增, 独特引物组合会因个体或物种所包含的内含子、启动子及间隔区间的不同而产生多态性[6]。在反应体系优化过程中, SRAP-PCR会因为不同的反应条件、反应程序及物种差异, 会产生不同的扩增结果, 体系中每一个因素的变化都会对导致扩增结果偏差[29]。本研究首先利用单因素试验确定反应体系中5大因素的浓度范围, 以减少盲目性[30]。依据正交试验设计再次进行多因素多水平综合检测, 充分发挥了正交试验的均衡分散、综合可比的特点, 快速有效地明确各因素的组合关系[31]。同时选择直接观察方法与新复极差法对扩增反应中各因素的影响作用及各因素最优浓度进行综合分析评估, 削弱或避免了仅仅依靠主观判断的误差[32]。本研究中, 两次正交试验重复评分差别不明显, 此结果与陈丽君等[33]对苦楝的研究结果一致, 证明SRAP-PCR反应体系相对比较稳定。经新复极差法研究发现, 5大因素中Mg2+ 的极差R最大, 说明在体系中Mg2+ 影响力最大。作为Taq DNA聚合酶的激活剂, Mg2+ 直接影响扩增过程中的酶活性。若体系中Mg2+ 浓度过高, 会直接导致非特异性扩增的发生, 但为了保证正常激活酶活性, 浓度不能过少, 否则会得到不理想的扩增结果, 影响扩增结果的准确性和重复性。在本研究中, Mg2+ 在体系中具有最重要的作用, 后续的试验中一定要着重注意该因素的一致性。起到次要作用的因素为Taq DNA聚合酶。Taq DNA酶浓度必须在一个合适范围内才能成功地扩增出特异性片段, 浓度少, 无法得到准确数量的扩增片段, 浓度大, 会扩增出多余的非特异性片段。引物的浓度同样是PCR扩增过程中的重要因素, 若出现模板DNA无法与之配对, 扩增失败的结果, 则与浓度过低有关; 产生非特异性性片段和形成引物二聚体, 则可能是引物浓度过高造成的。dNTP是PCR扩增反应的底物, 若扩增产率降低或聚合酶渗入则与其有关; 模板DNA是扩增反应的基础, 但如果DNA质量较高, 适量的浓度一般不会对扩增结果产生过多的影响[34-35]。各物种所含基因的差异及PCR试验的外在条件, 都会直接或间接的影响到扩增结果[18], 因此, 不同的物种应具有独立稳定的反应条件及体系。
在进行引物能力评估时, 可依据PIC值对引物进行初步判断。当PIC > 0.5, 表示该引物或引物组合所呈现的多态性最好, 贡献率较高, 可以最大程度的反应遗传多样性。当0.25 < PIC < 0.5, 或者PIC值< 0.25时, 则分别说明该引物扩增得到的多态位点处于中等或较低水平[36-37]。本研究中30对SRAP引物组合的PIC平均值为0.41, 说明SRAP分子标记的引物组合可以很好地反映引物的区分能力, 能有效准确度地揭示红椿不同种源的遗传多样性。
针对含有大量次生代谢产物的母树叶片, 本研究采用了有效的处理方法, 保证了DNA的提取质量; 采用多种检测手段进行SRAP分子标记体系优化, 并获得最优反应体系, 弥补了单一优化选择手段的不足, 区别了各因素在体系中的影响作用。以上均为其他物种进行DNA提取及SRAP分子标记提供参考依据。
-
本研究成功地获得红椿SRAP分子标记的最优体系, 体系如下: 25 μL体系中, 模板DNA浓度为25 ng, 引物浓度为0.3 μmol·L-1(正反引物浓度相同)、Taq DNA聚合酶为1 U、Mg2+ 浓度为2.5 mmol·L-1及dNTP浓度为0.3 mmol·L-1, 2.5 μL 10×Buffer, 重蒸水补齐体系。通过不同种源红椿基因组DNA的重复验证, 同样获得了稳定清晰、多态性较强的扩增条带, 表明所确定的最优体系稳定可靠, 适用性较强。利用最优体系进行红椿SRAP分子标记的引物筛选, 从1 505对引物组合中筛选出30对多态性高, 重复性强的引物组合, 用于红椿后续遗传多样性研究中。
红椿SRAP反应体系优化及引物筛选
Optimization and Primers Selection of SRAP-PCR System in Toona ciliata Roem.
-
摘要:
目的 优化相关序列扩增多态性(SRAP)体系内的不同组分,建立适用于红椿SRAP分子标记的反应体系,并进一步从SRAP引物组合中筛选出稳定、多态性好的引物组合,为红椿遗传多样性研究奠定试验基础。 方法 针对SRAP-PCR反应体系中5个因素各设置8个水平,先利用单因素试验确定浓度梯度,后在确定的梯度范围内选定4个水平,按照正交试验L16(45)进行优化,结合正交直观分析法和新复极差法对各因素进行优化筛选。 结果 确定最优体系为总体系25 μL,模板DNA 25 ng,上下游引物各0.3 μmol·L-1,Taq DNA聚合酶1U,Mg2+ 2.5 mmol·L-1,dNTP 0.3 mmol·L-1。利用稳定的SRAP-PCR体系,从1 505对SRAP引物组合中筛选出30对优质引物组合。 结论 通过不同种源红椿基因组DNA的重复验证,获得了稳定清晰、多态性较强的扩增条带,表明所确定的最优体系稳定可靠,适用性较强,可用于不同种源红椿遗传多样性研究的后续实验。 Abstract:Objective To optimize the different components of sequence-related amplified polymorphism (SRAP)and to establish suitable SRAP-PCR system forToona ciliata Roem., and to select high-stability polymorphic band SRAP primer combinations. Method The experiment basis for genetic diversity of T. ciliate was established. Five factors each with eight concentration levels were screened to the suitable concentration range in the PCR reaction system using single factor experiment. After that, four levels were selected in each range of the five factors. The orthogonal experiment of L16 (45)was carried out for optimization. Combining with orthogonal design-direct analysis and SSR to optimize these factors and the optimized system was determined. Result The optimal SRAP-PCR system was established, containing 25 ng template DNA, 0.3 mmol·L-1 each dNTP, 0.3 μmol·L-1 each primer, 2.5 mmol·L-1 each Mg2+ and 1 U Taq DNA polymerase in a total volume of 25 μL. 30 primer combinations were selected from 1 505 s using the stability of the PCR system. Conclusion Through repeated validations using DNA of different T. ciliata provenances, the clear polymorphic bands were obtained. The result showed that the optimal SRAP-PCR system can be used for the follow-up experiments on T. ciliata genetic diversity research in virtue of its stable, reliable, and good applicability. -
Key words:
- Toona ciliata Roem.
- / SRAP-PCR
- / reaction system
- / optimization
-
表 1 红椿SRAP-PCR体系优化试验材料
Table 1. Experimental material of T. ciliata for optimizing SRAP-PCR system
编号
No.种源代码
provenance省份
province纬度
latitude/N经度
longitude/E海拔
altitudeP1 乐昌 广东 25°07' 113°20' 359 P2 黄山 安徽 30°16' 118°08' 499 P3 望谟 贵州 25°10' 106°05' 500 P4 永仁 云南 25°01' 101°32' 1 539 P5 宜丰 江西 27°19' 115°26' 337 P6 城步 湖南 27°14' 111°28' 737 P7 南平 福建 26°38' 118°10' 800 P8 会东 四川 27°23' 102°09' 1 862 表 2 正交试验各因子水平设计
Table 2. The design of factors and the levels for orthogonal experimental
因子
factor水平level 1 2 3 4 Taq酶/U 1 1.25 1.5 1.75 Mg2+/(mmol·L-1) 1.5 2 2.5 3 dNTP/(mmol·L-1) 0.15 0.2 0.25 0.3 引物Primer/(μmol·L-1) 0.2 0.3 0.4 0.5 DNA模板/ng 25 50 75 100 表 3 SRAP-PCR正交试验设计[L16(45)]
Table 3. L16(45)orthogonal experimental of SRAP-PCR
处理编号 Taq DNA酶/(U) Mg2+/(mmol·L-1) dNTP/(mmol·L-1) 引物/(μmol·L-1) DNA模板/ng 1 1 1.5 0.15 0.2 25 2 1 2 0.2 0.3 50 3 1 2.5 0.25 0.4 75 4 1 3 0.3 0.5 100 5 1.25 1.5 0.2 0.4 100 6 1.25 2 0.15 0.5 75 7 1.25 2.5 0.3 0.2 50 8 1.25 3 0.25 0.3 25 9 1.5 1.5 0.25 0.5 50 10 1.5 2 0.3 0.4 25 11 1.5 2.5 0.15 0.3 100 12 1.5 3 0.2 0.2 75 13 1.75 1.5 0.3 0.3 75 14 1.75 2 0.25 0.2 100 15 1.75 2.5 0.2 0.5 25 16 1.75 3 0.15 0.4 50 表 4 不同处理得分情况表
Table 4. The result of range analysis for the factors
处理treatment 重复1 copy 1 重复2 copy 2 均值mean 1 6 7 6.5 2 15 14 14.5 3 13 13 13 4 14 15 14.5 5 1 2 1.5 6 2 1 1.5 7 9 9 9 8 12 12 12 9 3 3 3 10 11 11 11 11 16 16 16 12 10 10 10 13 7 6 6.5 14 5 5 5 15 8 8 8 16 4 4 4 表 5 各因素极差分析结果
Table 5. The result of range analysis for the factors
水平
levelTaq DNA聚合酶 Mg2+ dNTP 引物primer 模板DNA浓度
DNA concentration总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 总分sum 平均分mean 1 48.5 12.125 17.5 4.375 28 7 30.5 7.625 37.5 9.375 2 24 6 32 8 34 8.5 49 12.25 30.5 7.625 3 40 10 46 11.5 33 8.25 29.5 7.375 31 7.75 4 23.5 5.875 40.5 10.125 41 10.25 27 6.75 37 9.25 极差range 25 6.25 28.5 7.125 13 3.25 22 5.5 7 1.75 表 6 30对SRAP引物的总条带数、多态性条带数、多态性比率及PIC值
Table 6. Total number of bands, polymorphic bands, polymorphism and polymorphic information content (PIC) obtained from 30 SRAP primer combinations
引物组合primer 总条带数 多态性条带 多态百分率/% PIC值 Me13Em8 22 15 68.18 0.37 Me14Em6 23 17 73.91 0.40 Me17Em21 27 21 77.78 0.38 Me19Em13 25 21 84.00 0.42 Me19Em15 26 23 88.46 0.42 Me19Em18 17 11 64.71 0.41 Me19Em27 25 21 84.00 0.41 Me22Em21 26 21 80.77 0.41 Me22Em27 28 20 71.43 0.41 Me23Em22 30 26 86.67 0.44 Me23Em31 28 21 75.00 0.41 Me27Em7 25 21 84.00 0.44 Me27Em18 40 31 77.5 0.39 Me27Em28 25 21 84.00 0.39 Me28Em18 36 27 75.00 0.40 Me31Em24 36 29 80.56 0.43 Me32Em17 28 20 71.43 0.43 Me32Em26 32 28 87.50 0.42 Me33Em27 23 11 47.83 0.35 Me34Em13 25 19 76.00 0.39 Me35Em13 27 15 55.56 0.42 Me35Em14 26 15 57.69 0.40 Me35Em32 27 14 51.85 0.45 Me38Em21 24 19 79.17 0.39 Me38Em24 29 21 72.41 0.42 Me38Em32 28 26 92.88 0.42 Me39Em32 19 18 94.74 0.43 Me40Em13 28 23 82.14 0.42 Me40Em22 21 15 71.43 0.33 Me43Em19 33 26 78.79 0.43 合计Total 809 616 平均值 Mean 26.97 20.53 75.85 0.41 -
[1] Neil J, Helen O H T. Markers and mapping revisited: finding your gene[J]. New Phytologist, 2009, 183: 935-966. doi: 10.1111/j.1469-8137.2009.02933.x [2] Agarwal M, Shrivastava N, Padh H. Advances in molecular marker techniques and their applications in plant sciences[J]. Plant Cell Rep, 2008(27): 617-631. [3] Li G, Quiros C F. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. TAG Theoretical and Applied Genetics, 2001, 103(2): 455-461. [4] Sun S, Gao W, Lin S, et al. Analysis of genetic diversity in Ganoderma population with a novel molecular marker SRAP[J]. Applied Microbiology and Biotechnology, 2006, 72(3): 537-543. doi: 10.1007/s00253-005-0299-9 [5] Feng N, Xue Q, Guo Q, et al. Genetic Diversity and Population Structure of Celosia argentea and Related Species Revealed by SRAP[J]. Biochemical Genetics, 2009, 47(7-8): 521-532. doi: 10.1007/s10528-009-9257-z [6] Ren N, Liu J, Yang D, et al. Sequence-related amplified polymorphism(SRAP)marker as a new method for identification of endophytic fungi from Taxus[J]. World Journal of Microbiology and Biotechnology, 2012, 28(1): 215-221. doi: 10.1007/s11274-011-0810-7 [7] Yin Y, Liu Y, Li H, et al. Genetic diversity of Pleurotus pulmonarius revealed by RAPD, ISSR, and SRAP fingerprinting[J]. Curr Microbiol, 2014, 68(3): 397-403. doi: 10.1007/s00284-013-0489-0 [8] Youssef M, James A C, Rivera-Madrid R, et al. Musa genetic diversity revealed by SRAP and AFLP[J]. Mol Biotechnol, 2011, 47(3): 189-199. doi: 10.1007/s12033-010-9328-8 [9] Li P, Zhan X, Que Q, et al. Genetic Diversity and Population Structure of Toona Ciliata Roem. Based on Sequence-Related Amplified Polymorphism(SRAP)Markers[J]. Forests, 2015, 6(4): 1094-1106. [10] 龙汉利, 冯毅, 向青, 等. 四川盆周山地红椿生长特性研究[J]. 四川林业科技, 2011(03): 37-41. doi: 10.3969/j.issn.1003-5508.2011.03.006 [11] Boland D J. Toona ciliata M. Roemer. [M]. Wallingford: CAB International, 2000: 6472-6485. [12] Zacaroni A B, Pozza E A, Mansur T D O F, et al. Occurrence of Phyllachora balansae in Toona ciliata in Southern Minas Gerais State, Brazil. [J]. Summa Phytopathologica, 2013, 39(3): 219-221. doi: 10.1590/S0100-54052013000300018 [13] 刘均利, 杨柳璐, 刘青, 等. 红椿的组织培养与植株再生[J]. 林业科技, 2014(06): 1-5. [14] 文亚峰, 韩文军, 吴顺. 植物遗传多样性及其影响因素[J]. 中南林业科技大学学报, 2010(12): 80-87. doi: 10.3969/j.issn.1673-923X.2010.12.016 [15] Zeng J, Zou Y, Bai J, et al. Preparation of Total DNA from " Recalcitrant Plant Taxa[J]. Acta Botanica Sinica, 2002, 44(6): 694-697. [16] 李建光, 李荣生, 李发根, 等. 蛇皮果基因组DNA提取及其RAPD条件优化[J]. 中南林业科技大学学报, 2012(06): 110-114. [17] 令狐斌, 侯思宇, 孙朝霞, 等. 苦荞SRAP分子标记体系优化与遗传多样性分析[J]. 中国农业大学学报, 2015(01): 37-43. [18] 糜亚男, 张水寒, 蔡媛, 等. 杜仲SSR-PCR反应体系的优化[J]. 中国实验方剂学杂志, 2015(02): 1-6. [19] 武正前, 李雪虎, 梁剑平, 等. 大青叶SRAP-PCR反应体系的建立与优化研究[J]. 植物研究, 2015(02): 310-314. [20] Alghamdi S, Al-Faifi S, Migdadi H, et al. Molecular Diversity Assessment Using Sequence Related Amplified Polymorphism(SRAP)Markers in Vicia faba L. [J]. International Journal of Molecular Sciences, 2012, 13(12): 16457-16471. doi: 10.3390/ijms131216457 [21] Aneja B, Yadav N R, Chawla V, et al. Sequence-related amplified polymorphism(SRAP)molecular marker system and its applications in crop improvement[J]. Molecular Breeding, 2012, 30(4): 1635-1648. doi: 10.1007/s11032-012-9747-2 [22] Huang C, Liu G, Bai C, et al. Genetic Analysis of 430 Chinese Cynodon dactylon Accessions Using Sequence-Related Amplified Polymorphism Markers[J]. International Journal of Molecular Sciences, 2014, 15(10): 19134-19146. doi: 10.3390/ijms151019134 [23] Jing Z B, Wang X P, Cheng J M. Analysis of genetic diversity among Chinese wild Vitis species revealed with SSR and SRAP markers[J]. Genet Mol Res, 2013, 12(2): 1962-1973. doi: 10.4238/2013.June.13.5 [24] Mokhtari N, Rahimmalek M, Talebi M, et al. Assessment of genetic diversity among and within Carthamus species using sequence-related amplified polymorphism(SRAP)markers[J]. Plant Systematics and Evolution, 2013, 299(7): 1285-1294. doi: 10.1007/s00606-013-0796-8 [25] Polat I, Kacar Y A, Yesiloglu T, et al. Molecular characterization of sour orange(Citrus aurantium)accessions and their relatives using SSR and SRAP markers[J]. Genet Mol Res, 2012, 11(3): 3267-3276. doi: 10.4238/2012.September.12.10 [26] 赵秀娟, 宋建文, 胡开林. 苦瓜种质遗传多样性的SRAP标记分析[J]. 植物遗传资源学报, 2013(01): 78-84. doi: 10.3969/j.issn.1672-1810.2013.01.012 [27] Shen J, Jia X, Ni H, et al. AFLP analysis of genetic diversity of Jatropha curcas grown in Hainan, China[J]. Trees. 2010, 24(3): 455-462. doi: 10.1007/s00468-010-0413-1 [28] Brunt C, Read J, Sanson G D. Changes in resource concentration and defence during leaf development in a tough-leaved(Nothofagus moorei)and soft-leaved(Toona ciliata)species[J]. Oecologia, 2006, 148(4): 583-592. doi: 10.1007/s00442-006-0369-4 [29] 李芳芳, 杨少宗, 柳新红, 等. 枫香树DNA提取及SRAP-PCR反应体系的建立与优化[J]. 河南农业大学学报, 2015(01): 46-51. [30] 刘明骞, 陈丽君, 欧阳昆唏, 等. 剑豆SRAP-PCR反应体系的建立及优化[J]. 华南农业大学学报, 2015(01): 75-78. [31] 王军, 杨荣萍, 洪明伟, 等. 石榴SPAR-PCR反应体系的正交设计优化及验证[J]. 云南农业大学学报: 自然科学, 2013(03): 376-379. doi: 10.3969/j.issn.1004-390X(n).2013.03.016 [32] 刘萌芽, 黄如葵, 黄玉辉, 等. 正交直观分析法和新复极差法优化苦瓜SRAP反应体系研究[J]. 北方园艺, 2014(05): 85-88. [33] 陈丽君, 刘明骞, 廖柏勇, 等. 苦楝SRAP-PCR反应体系的建立及优化[J]. 华南农业大学学报, 2015, 36(3): 104-108. [34] 陈罡, 关明东, 叶景丰, 等. 杨树SRAP-PCR反应体系的建立与优化[J]. 北方园艺, 2010(16): 132-134. [35] 何仁锋, 冯尚国, 陈喆, 等. 药用菊花SSR-PCR反应体系优化及引物筛选[J]. 分子植物育种, 2015(02): 367-378. [36] Wang B H, Zhu P, Yuan Y L, et al. Development of EST-SSR markers related to salt tolerance and their application in genetic diversity and evolution analysis in Gossypium[J]. Genet Mol Res, 2014, 13(2): 3732-3746. doi: 10.4238/2014.May.13.1 [37] Xie W, Zhang X, Cai H, et al. Genetic diversity analysis and transferability of cereal EST-SSR markers to orchardgrass(Dactylis glomerata L. )[J]. Biochemical Systematics and Ecology, 2010, 38(4): 740-749. doi: 10.1016/j.bse.2010.06.009 -




 下载:
下载: