-
穗花杉属(Amentotaxus)是红豆杉科(Taxaceae)的一个属,作为一个非常古老的家系存在于欧亚大陆。在第三纪和第四纪冰川时代,穗花杉属经历了多次群体扩张和紧缩,也因此形成了如今的分布格局[1]。穗花杉(Amentotaxus argotaenia (Hance) Pilger)是穗花杉属的一个种,有“冰川元老”的美称,主要分布于我国南部地区,以福建、江西、广东等省为主,也是我国三级重点保护的珍稀濒危植物。
在裸子植物系统分类上,穗花杉具有很重要的研究价值,因为研究者们从穗花杉被发现以来,一直对它的分类地位有争论[2]。早在1883年Hance发现穗花杉将其定名发表,并将其置于罗汉松属(Podocarpus)内[3]。20世纪初,Pilger根据穗花杉小孢子叶球形态,将它转入三尖杉属(Cephalotaxus)[4]。但是,随后Pilger根据穗花杉雄状花序的形态特征将穗花杉单独列为三尖杉科(Cephalotaxaceae)的一个属─穗花杉属[5]。1931年,通过观察穗花杉生殖器官的形成特征,工藤认为它应该成立一个穗花杉科或是作为红豆杉科的一个亚科或族[6]。1951年,Florin发现三尖杉属和穗花杉属间的气孔结构和大孢子叶球有差异,所以将穗花杉属归入红豆杉科。我国植物学者也在上世纪从各个学科领域对穗花杉的系统分类学做了大量的研究[7]。其中,马忠武发现穗花杉属植物不含有红豆杉属和榧树属的植物中存在的双黄酮类特征化学成分,所以他对穗花杉属归于红豆杉科提出怀疑[8]。
为了为穗花杉的研究提供基因层面的数据,本研究利用HiSeq2500技术对穗花杉茎叶进行了转录组测序。穗花杉转录组测序共得到约8.14 Gb的clean data,从头拼接后共获得82 884条Unigene和140 286条Transcript。通过功能注释,得到了萜类合成相关基因,以及紫杉二烯合成酶的同源基因。同时预测了穗花杉转录组序列的SSR位点。这为今后穗花杉萜类次生代谢和穗花杉分类学地位的研究奠定了一定的基础。
-
穗花杉材料由桂林小庐山生态农业发展有限公司提供。盆栽于中国林业科学研究院温室里。取当年生单株带叶的枝条,采集后立即放入液氮中,之后存于-80℃冰箱中待用。
-
总RNA的提取采用Omega Total RNA kit II (Omega, USA)试剂盒,按照实验说明书操作。分别采用Nanodrop、Qubit 2.0、Aglient 2100方法检测RNA样品的纯度、浓度和完整性。样品的RIN值>7。总RNA检测合格后,用带有Oligo(dT)的磁珠富集mRNA,再将mRNA随机打断成小片段。以片段化的mRNA为模板,合成双链cDNA,纯化后进行末端修复、加A尾和接头。经过片段大小筛选后,通过PCR扩增富集得到cDNA文库。构建好的文库利用Illumina HiSeq2500进行测序。
-
得到测序数据后,对raw read进行数据过滤,去除其中的接头序列及低质量read获得高质量的clean read(质量值Q < 20的碱基数占整个read的50%以上)。得到的clean read利用Trinity[9]进行从头拼接成为片段(contig),参数选用Trintity的省缺参数Kmer =25。然后,拼接好的片段进一步整合成为转录本和unigene。
-
使用BLASTx软件将拼接得到的unigene序列与NR、Swiss-Prot、GO、COG、KOG、KEGG数据库比对,使用KOBAS2.0[10]得到unigene在KEGG中的KEGG Orthology结果,预测完unigene的氨基酸序列之后使用HMMER[11]软件与Pfam[12]数据库比对,获得unigene的注释信息。
-
使用MISA软件分析穗花杉unigene序列可以得到6种类型的SSR:单碱基重复SSR、双碱基重复SSR、三碱基重复SSR、四碱基重复SSR、五碱基重复SSR、六碱基重复SSR。这六种类型SSR设置的参数分别是重复10、6、5、5、5和5次。
-
利用欧洲红豆杉(Taxus baccata L.)紫杉醇生物合成关键酶-紫杉二烯合成酶(Taxadiene synthase gene, TbTS)蛋白质的氨基酸序列对本地穗花杉转录组进行TBLASTN分析[13]。比对参数为E值< 1e-25。
-
利用Illumina HiSeq2500技术对穗花杉茎叶进行转录组测序后,获得了32 321 978个reads片段,包含了8 141 132 186个核苷酸序列信息。质量值Q≥30碱基数占所有read的碱基数的比例即Q30%为89.91%。穗花杉转录组测序共得到约8.14 Gb的clean data,组装得到4 898 457条序列重叠群(Contig),总长度约为261 826 164 bp,平均长度及N50分别为53.45 bp和48 bp。进一步对Contigs进行拼接后,共获得82 884条Unigene序列和140 286条Transcript序列,总长度为分别为55 775 850 bp和143 129 259 bp,平均长度为673 bp和1 020 bp,N50的长度为1 314 bp和1 932 bp。长度大于1 000 bp的Unigene和Transcript序列分别占到18.14%和34.39%(表 1)。
表 1 穗花杉转录组从头组装结果统计
Table 1. The statistical result of de novo assembly of A. argotaenia transcriptome
Length Range Contig Transcript Unigene 200~300 43 871(0.90%) 43 593(31.07%) 37 524(45.27%) 300~500 22 709(0.46%) 26 027(18.55%) 19 081(23.02%) 500~1 000 13 501(0.28%) 22 426(15.99%) 11 247(13.57%) 1 000~2 000 9 636(0.20%) 25 925(18.48%) 8 724(10.53%) 2 000+ 4 045(0.08%) 22 315(15.91%) 6 308(7.61%) Total Number 4 898 457 140 286 82 884 Total Length 261 826 164 143 129 259 55 775 850 N50 Length 48 1 932 1 314 Mean Length 53.45 1 020 673 -
为了能够直观地研究穗花杉转录组信息,将所拼接得到的82 884条unigene分别与NR、Swiss-Prot、GO、COG、KOG、KEGG数据库进行比对分析。结果显示,共有27 495条unigene至少被一个数据库注释,占unigene总数的30.14%(表 2)。
表 2 穗花杉unigene功能注释统计
Table 2. The statistical result of functional annotation of A. argotaenia unigenes
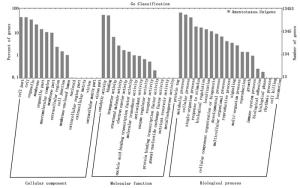
Anno_Database Annotated_Number 300≤length < 1 000 length≥1 000 COG_Annotation 8 206 1 673 5 517 GO_Annotation 13 453 3 726 7 154 KEGG_Annotation 5 145 1 154 3 330 KOG_Annotation 15 694 4 590 8 056 Pfam_Annotation 16 816 4 124 10 810 Swissprot_Annotation 17 575 5 299 9 464 nr_Annotation 26 996 8 499 13 002 All_Annotated 27 495 8 704 13 037 利用COG数据库对得到的82 884条穗花杉unigene进行功能注释,其中被注释到的unigene一共有8 206条,分别被注释到24个COG分类中。其中,“一般功能基因”(General function prediction only)、“复制、重组和修复”(Replication, recombination and repair)和“转录”(Transcription)是最大的3个类群,分别有2 141、1 534和951条unigene。然而,“核结构”(Nuclear structure)是最小的类群,只有1条unigene(图 1)。

图 1 穗花杉转录组Unigene的COG功能聚类
Figure 1. COG function classification of the unigenes in A. argotaenia transcriptome
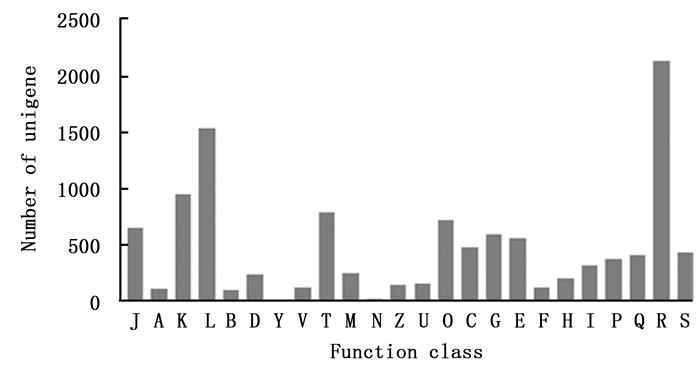
对82 884条unigene进行GO功能注释表明,其中有13 453条unigene分别注释到51个功能组,归纳为生物学过程(Biological Process)、细胞组分(Cellular Component)和分子功能(Molecular Function)三大部分,分别含有20、16及15个功能组。在生物学过程部分中,差异表达基因在代谢过程(metabolic process)和细胞过程(cellular process)功能组中注释的unigene数目最多,分别为8 846和7 206条;在细胞组分部分中,细胞部分(cell part)和细胞(cell)功能组差异基因数目最多,分别为5 609和5 551条;在分子功能部分中,催化活性(catalytic activity)和结合(binding)功能组分别含有6 989和6 676条(图 2)。
为了能够把穗花杉的基因信息作为一个整体的网络进行研究,我们利用KEGG数据库对穗花杉转录组进行分析。其中,代谢途径(ko01100)和次生代谢生物合成(ko01110)中的unigene数最多,分别为1 477和664条,这为查找特定次生代谢途径中的基因提供了很好的基础。
-
利用MISA软件搜索穗花杉的15 032条Unigene,共在2 420条unigene中搜索到2 827个SSRs位点。具有1个以上SSRs的unigene是339条。单核苷酸重复SSR出现频率最高(60.1%),其次为三核苷酸(25.4%)和二核苷酸(12.5%)。其它三种类型的都很少:四核苷酸(0.9%)、五核苷酸(0.6%)、六核苷酸(0.5%)(表 3)。
表 3 穗花杉SSR位点分析
Table 3. The analysis of SSR site of A. argotaenia
Motif Length Repeat Numbers Total % 5 6 7 8 9 10 >10 Mono - - - - - 894 805 1 699 60.1 Di - 191 71 29 20 16 25 352 12.5 Tri 464 160 70 21 2 - 1 718 25.4 Tetra 22 3 - 1 - - - 26 0.9 Penta 15 1 1 - - - - 17 0.6 Hexa 7 4 3 1 - - - 36 0.5 Total 508 359 145 52 22 910 831 2 827 % 18.0 12.7 5.1 1.8 0.8 32.2 29.4 -
利用KEGG通路中的基因信息查找被KEGG数据库注释的穗花杉unigene,获得牦牛儿基焦磷酸合成酶(GPS)、法尼基焦磷酸合成酶(FPS)和牦牛儿基牦牛儿基焦磷酸合成酶(GGPS)的同源基因,分别为1、1和5个(图 3)。利用紫杉二烯合成酶TbTS基因序列对穗花杉本地转录组进行BLAST分析,得到13个同源基因,这为今后进一步验证穗花杉中是否存在紫杉二烯合成酶提供了基因基础(图 3)。
-
树木的基因组较大,测一个基因组需要较高的成本。而且树木的染色体构型较为复杂,除了成本问题,在染色体拼接方面也存在巨大的挑战。随着测序技术的发展,转录组测序已经越来越普遍,也为进一步探测基因组研究缺乏物种功能基因组变化的网络模式提供条件[14]。在红豆杉科中,红豆杉属、榧树属中的香榧都有转录组测序[15-16]。红豆杉属中的物种不仅有组织和细胞系的转录组测序,还有激素处理后的转录组测序[17]。然而,穗花杉属中的穗花杉的基因数据比较匮乏,本研究利用HiSeq2500对穗花杉的茎叶混合样品进行转录组测序,共获得82 884条unigene和140 286条transcript。其中,有27 495条unigene得到注释,研究结果为穗花杉的遗传基础研究提供了基因层面的数据。
穗花杉在分类学研究上具有很高的学术价值。自发现以来,它的分类归属一直是植物学家争论的问题。研究者从植物形态学、胚胎学、解剖学、孢粉学和植物化学等方面对穗花杉的系统分类地位进行了广泛的研究。多数学者认为穗花杉具有红豆杉科某些共同特征,与三尖杉科关系密切,是红豆杉与三尖杉科之间联系的桥梁[2]。但是,少有研究者从分子遗传层面研究穗花杉的系统分类地位。仅有管启良等[18]对穗花杉染色体层面对穗花杉的系统分类地位进行了研究,得出结论归于罗汉松科。本研究利用高通量测序技术对穗花杉转录组测序,得到大量基因数据,为以后深入研究穗花杉的系统分类打下基础。
穗花杉属缺乏足够的遗传背景,Ge等[1]只利用ISSR技术对穗花杉属的各个种之间的遗传多样性和群体结构进行分析。虽然ISSR引物通用性好,但是得到的多态性位点有限。在本研究中,我们在穗花杉转录组数据中一共预测到2 827个SSR位点,远远高于ISSR通用引物数,为以后SSR位点的开发提供了遗传基础。SSR位点的多态性也为生物遗传多样性的研究提供了一个重要的手段。
紫杉醇是一个重要的萜类化合物,最早是在紫杉树皮中发现的。穗花杉属是红豆杉科的一个属,是否能够合成紫杉醇依然未知。根据植物化学系统学概念,在红豆杉科中的穗花杉属植物里面也可能具有紫杉醇。穗花杉转录组功能注释得到5个GGPS基因,还有20个跟二萜生物合成通路基因。紫杉二烯合酶是紫杉醇合成过程中一个重要的限速酶,只要穗花杉中存在紫杉二烯合酶(Taxadiene synthease, TS)基因,那么就为证明穗花杉可以合成紫杉醇提供了重要的证据。对本地穗花杉转录组数据进行TBLASTN分析, 预测到的30个unigene。为以后的验证穗花杉是否能够合成紫杉醇提供了候选基因。
-
本研究通过穗花杉转录组测序共获得82 884条unigene,有27 495条unigene得到注释,其中有近20个unigene与萜类合成途径有关,为以后该物种萜类合成途径研究,特别是紫杉二烯合成酶编码基因的研究打下了基础,也为穗花杉系统分类地位的研究提供新的遗传信息。另外,在穗花杉转录组数据中,我们一共预测到2 827个SSR位点,这为今后穗花杉SSR位点的开发与遗传多样性评估提供了有用的分子标记。
穗花杉的转录组测序及其转录组特性分析
Transcriptome Sequencing and Analysis of Amentotaxus argotaenia (Hance) Pilger
-
摘要:
目的 通过对红豆杉科穗花杉属的穗花杉进行转录组测序,为穗花杉的萜类合成途径和分类学研究提供支持。 方法 利用HiSeq2500技术对穗花杉的茎叶进行转录组测序。 结果 穗花杉转录组测序共得到8.14 Gb的clean data。从头组装共获得82 884条unigene,其中有27 495条unigene被注释。此外,在82 884条unigene中共搜索到2 827个SSR位点,单核苷酸重复SSR出现频率最高(60.1%),其次为三核苷酸重复SSR(25.4%)。在穗花杉unigene中挖掘出了1个牦牛儿基焦磷酸合成酶(GPS)、1个法尼烯基焦磷酸合成酶(FPS)和5个牦牛儿基牦牛儿基焦磷酸合成酶(GGPS)的同源基因,同时得到紫杉二烯合成酶(TS)同源基因13个。 结论 发现了20个与萜类合成有关的unigene和2 827个SSR位点,为今后穗花杉萜类生物合成研究,特别是紫杉二烯合成酶编码基因的研究打下了基础,也为该物种系统分类地位及遗传多样性研究提供了新的遗传信息。 Abstract:Objective The transcriptome of Amentotaxus argotaenia (Hance) Pilger of Amentotaxus in Taxaceae was sequenced and analyzed for its terpenoid biosynthetic pathway and taxonomy research. Method The transcriptome of the mixture of stem and leave of A. argotaenia was sequenced by using HiSeq2500. Result 8.14 Gb clean data was obtained from the transcriptome of A. argotaenia. 82 884 unigene were obtained and 27 495 unigene were annotated using the six public databases. Besides, 2 827 SSRs were identified from the 82 884 unigene. The most abundant type of repeat motif was mono-nucleotides (60.1%), followed by tri-nucleotides (25.4%). Among the unigenes of A. argotaenia, one homologous gene of geranyl diphosphate synthase (GPS), one homologous gene of farnesyl diphosphate synthase (FPS) and five homologous genes of geranylgeranyl diphosphate synthase (GGPS) were identified. In addition, 13 homologous genes to taxadiene synthase gene (TS) in Taxus were obtained using TBLASTN. Conclusion In this study, 20 unigenes relating to terpenoid biosynthesis and 2 827 SSRs were identified in A. argotaenia. Our work will facilitate the study about the terpenoid biosynthetic genes especially taxadiene synthase gene in A. argotaenia and may provide some foundational genetic data to study its taxonomy and diversity. -
Key words:
- Amentotaxus argotaenia
- / transtriptome
- / SSR
- / terpenoid biosynthesis pathway gene
- / taxonomy
-
表 1 穗花杉转录组从头组装结果统计
Table 1. The statistical result of de novo assembly of A. argotaenia transcriptome
Length Range Contig Transcript Unigene 200~300 43 871(0.90%) 43 593(31.07%) 37 524(45.27%) 300~500 22 709(0.46%) 26 027(18.55%) 19 081(23.02%) 500~1 000 13 501(0.28%) 22 426(15.99%) 11 247(13.57%) 1 000~2 000 9 636(0.20%) 25 925(18.48%) 8 724(10.53%) 2 000+ 4 045(0.08%) 22 315(15.91%) 6 308(7.61%) Total Number 4 898 457 140 286 82 884 Total Length 261 826 164 143 129 259 55 775 850 N50 Length 48 1 932 1 314 Mean Length 53.45 1 020 673 表 2 穗花杉unigene功能注释统计
Table 2. The statistical result of functional annotation of A. argotaenia unigenes
Anno_Database Annotated_Number 300≤length < 1 000 length≥1 000 COG_Annotation 8 206 1 673 5 517 GO_Annotation 13 453 3 726 7 154 KEGG_Annotation 5 145 1 154 3 330 KOG_Annotation 15 694 4 590 8 056 Pfam_Annotation 16 816 4 124 10 810 Swissprot_Annotation 17 575 5 299 9 464 nr_Annotation 26 996 8 499 13 002 All_Annotated 27 495 8 704 13 037 表 3 穗花杉SSR位点分析
Table 3. The analysis of SSR site of A. argotaenia
Motif Length Repeat Numbers Total % 5 6 7 8 9 10 >10 Mono - - - - - 894 805 1 699 60.1 Di - 191 71 29 20 16 25 352 12.5 Tri 464 160 70 21 2 - 1 718 25.4 Tetra 22 3 - 1 - - - 26 0.9 Penta 15 1 1 - - - - 17 0.6 Hexa 7 4 3 1 - - - 36 0.5 Total 508 359 145 52 22 910 831 2 827 % 18.0 12.7 5.1 1.8 0.8 32.2 29.4 -
[1] Ge X J, Zhou X L, Li Z C, et al. Low genetic diversity and significant population structuring in the relict Amentotaxus argotaenia complex (Taxaceae) based on ISSR fingerprinting[J]. Journal of Plant Research, 2005, 118(6): 415-422. doi: 10.1007/s10265-005-0235-1 [2] 何飞, 刘兴良, 马钦彦, 等. 珍稀濒危植物穗花杉的研究进展[J]. 四川林业科技, 2007, 28(6): 31-38. doi: 10.3969/j.issn.1003-5508.2007.06.006 [3] 孙同兴, 林金星. 中国特有植物穗花杉的生物学特性及其保护[J]. 广西植物, 1996, 16(4): 353-358. [4] Pilger R. Taxaceae in Engler's[J]. Das Pflanzenreich, 1903. [5] Pilger R. Gymnospermae in Engler A. and Prantl K[M]. Die Natrill Pflanzenfamilien. 2nd, 1926, 13: Leipzeg. [6] Kudo Y, Yamamoto Y. Amentotaxaceae[J]. Mater. Fl. Formosa IV Soc Trop Agric, 1931, 3: 110-111. [7] Florin R. Evolution in cordaites and conifers[J]. Acta Horti Bergiani, 1951, 15: 285-388. [8] 马忠武, 何关福, 印万芬. 双黄酮成分在红豆杉科各属、种中的分布[J]. 中国科学院大学学报, 1985, 23(3): 192-195. [9] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652. doi: 10.1038/nbt.1883 [10] Xie C, Mao X, Huang J, et al. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 2011, 39(suppl 2): W316-W322. [11] Eddy S R. Profile hidden Markov models[J]. Bioinformatics, 1998, 14(9): 755-763. doi: 10.1093/bioinformatics/14.9.755 [12] Finn R D, Baterman A, Clements J, et al[M]. Nucleic Acids Research, 2014, 42: D222-230. [13] Köksal M, Jin Y, Coates R M, et al. Taxadiene synthase structure and evolution of modular architecture in terpene biosynthesis[J]. Nature, 2011, 469(7328): 116-120. doi: 10.1038/nature09628 [14] Leung R K K, Dong Z Q, Sa F, et al. Quick, sensitive and specific detection and evaluation of quantification of minor variants by high-throughput sequencing[J]. Molecular BioSystems, 2014, 10(2): 206-214. doi: 10.1039/C3MB70334G [15] Hao D C, Ge G B, Xiao P G, et al. The first insight into the tissue specific taxus transcriptome via Illumina second generation sequencing[J]. PLoS One, 2011, 6(6): e21220. doi: 10.1371/journal.pone.0021220 [16] 易官美, 包燕春. 香榧转录组测序及生物信息学基础分析[J]. 山东农业大学学报: 自然科学版, 2016, 47(1): 19-24. doi: 10.3969/j.issn.1000-2324.2016.01.004 [17] Qiu D, Pan X, Wilson I W, et al. High throughput sequencing technology reveals that the taxoid elicitor methyl jasmonate regulates microRNA expression in Chinese yew (Taxus chinensis)[J]. Gene, 2009, 436(1): 37-44. [18] 管启良, 俞仲辂. 穗花杉染色体的研究[J]. 植物分类与资源学报, 1993(4): 385-391. -



 下载:
下载: