-
映山红(Rhododendron simsii Planch.),又名杜鹃花、山石榴,属于杜鹃花科(Ericaceae)杜鹃花属(Rhododendron)植物,具有极高的观赏价值和药用价值,是北温带森林生态系统中的主要成员[1-2]。研究映山红不同龄级种群的遗传多样性和遗传结构差异,探究影响因素,对映山红资源的可持续开发意义重大。
Wu等[3]采用AFLP显性标记对濒危物种大树杜鹃(R. protistum var. giganteum)的遗传多样性和遗传结构进行了研究。Liu等[4]采用ISSR和RAPD标记对长白山杜鹃(R. aureum Georgi)种群的遗传多样性和遗传结构进行了研究。基于随机引物,Kuttapetty等[5]发现树形杜鹃(R. arboreum)种群具有高的遗传多样性(Ht=0.21,Nm=1.13)。Zhao等[6]采用AFLP标记发现秀雅杜鹃(R. concinnum)种群在物种水平和种群水平上均有较高的遗传多样性。目前,对映山红的保护遗传学研究比较少,而且主要集中于空间尺度上,尚缺乏时间尺度上的研究。
微卫星标记(SSR)在基因组中广泛分布、多态性丰富、重复性好、特异性强、共显性遗传,在植物遗传多样性研究中被广泛运用,但是在映山红的遗传多样性研究中应用较少[7]。本研究在映山红空间遗传多样性研究的基础上,基于前期RNA-seq技术开发的SSR标记阐述世代结构类型丰富的大别山黄狮寨映山红种群时间尺度上的遗传多样性,揭示不同世代遗传多样性的变化规律,从分子层面探讨映山红的生存潜力和进化方向,为有效保护大别山野生映山红资源提供理论依据。
-
选取罗田县黄狮寨国营林场(31°10′14.63″ N,115°31′56.35″ E,海拔700~860 m)的映山红种群,该地属亚热带季风气候,年均气温为16.4℃,无霜期228天,年均降水量约1 400 mm,气候温和。千亩野生的映山红位于山坡的狭长地段。土层为弱酸性,水肥条件适中,透气性较好。
根据传统野外调查方法对黄狮寨映山红进行每株调查。参照李久林等[8]对马缨杜鹃提出的年龄级划分法,将映山红个体按照基径大小和分支数进行分组,基径>12.5 cm且分支数大于40个的为老树种群,基径为7.5~12.5 cm且分支数为20~25个的成树种群,基径2.5~7.5 cm且分支数为5~10个为小树种群,胸径 < 2.5 cm且独支的为幼苗种群。由于种群内幼苗和老树个体数量有限,因此4个种群各挑选15株大小比较一致的植株,采集当年生叶片,硅胶保存后带回实验室。
-
取嫩叶0.5 g,液氮冻存后,采用改良的CTAB法提取基因组DNA,用1%琼脂糖凝胶电泳检测总DNA的完整度[2]。紫外分光光度计检测DNA样品的浓度和纯度,稀释至50 ng· μL-1,于-20℃保存备用。
-
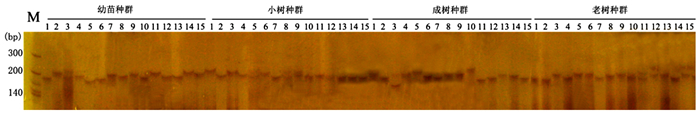
设置10 μL的SSR-PCR反应体系,包括5 μL的2×Taq PCR Mastermix,50 ng基因组DNA,正反向引物(20 mmol·L-1)各0.1 μL,灭菌的去离子水补足剩余体积。PCR扩增程序为94℃预变性10 min,然后进行35个扩增循环(94℃变性30 s,最适退火温度下退火40 s,72℃延伸40 s),最后增加一个7 min的72℃延伸阶段。PCR扩增产物在6%的变性聚丙烯酰胺凝胶上电泳,并用银染法染色。
-
依据20 bp DNA marker的电泳谱,判断扩增条带大小,采用人工读带方式记录SSR图谱,并按照凝胶上扩增产物的有无,构建“0/1”矩阵。利用POPGENE 32.0软件分析等位基因数(Na)、有效等位基因数(NE)、多态性位点比率(P)、观察杂合度(HO)、期望杂合度(HE)、Shannon多样性指数(I)、Nei基因多样性指数(h)、各龄级种群间的遗传距离(D)和遗传相似性度(S)、种群内近交系数(Fis)、总近交系数(Fit),以及遗传分化系数(Fst)、基因流(Nm)、Hardy-Weinberg平衡(HWE)等[9-10]。基于Nei’s遗传距离,采用MEGA 5.0软件进行UPGMA聚类分析[11]。
-
12个微卫星位点共检测到61个等位基因,每个位点为3!9个,等位基因大小为149~262 bp(表 1)。WRsE-37标记检测到9个等位基因(图 1),等位基因数最少的是WRsE-90和WRsE-97,仅检测到3个。在4个年龄级种群内分别检测到43、50、48、47个等位基因(表 2)。Shannon信息指数变化范围为1.025 0~1.188 5。Shannon信息指数反映的不同龄级映山红遗传多样性的顺序为:小树>成树>老树>幼苗。观察杂合度和期望杂合度的变化范围分别为0.676 4~0.881 2和0.607 7~0.690 7。Nei's基因多样度变化范围为0.578 2~0.654 4,平均为0.632 6,在小树种群内最大,最小的是幼苗种群,与Shannon信息指数反映的一致。4个种群均存在HO大于HE的情况,即杂合子过剩现象。经Bonferroni校正(P < 0.004),8个微卫星位点严重偏离Hardy-Weinberg平衡(表 3)。
表 1 微卫星标记信息
Table 1. Information of microsatellite markers
位点 Locus 正向引物(5′-3′)
Forward primer (5′-3′)反向引物(5′-3′)
Reverse primer (5′-3′)基序
Motif退火温度 Annealing temperature /℃ 片段大小
Size range /bpWRsE-37 CTTGCCACTTTGAGTTTGAG AGAGTAATTTGGAGGAAGCG (CT)28 55 200~212 WRsE-43 TGAACCCTTCTTCTTCTTCC TTTGATTGAAGGGTGGAGTG (TC)23 56 249~262 WRsE-70 TCTTCCGATTCCATCATTCC TGGGCGTGATTTGGTTATAA (CT)22 58 178~192 WRsE-77 TAGCTGCTTACTGTTGAAGG AGTAAAAGGGCTGAAACTGT (AG)22 57 149~165 WRsE-81 GCCCTATCCCTCAACTTTAC GAGGAGCGTGGTTAGTAATT (TC)21 56 229~253 WRsE-82 GTATGGGACCTGTGATTTCC CTCCAACTAGCTACTCCAAC (GA)24 57 228~236 WRsE-85 GAAATCTCGAATCACCTCCA AAGGTGTTGGTGGACTAATC (TC)21 56 149~167 WRsE-90 TTGAAGAACACTCAAGTTGC ACGTAGAACATTGCTTTCCT (GA)21 57 196~202 WRsE-93 GGTATCCGGTTTTCATCACT ATACCCACTAGCAACAGAGA (GA)23 57 233~256 WRsE-97 AGAAAACTGGGAGATGTGTC AGGTGATCATCTTTGCATGT (CT)21 58 222~246 WRsE-113 TATTGTACAGCTCCCCTTTG CCTCAATGTTCTATCGACGT (CT)23 57 185~207 WRsE-131 CTCTTTCCCCTTTCATGTGA AAGTGTTGAGTCCTCGTATG (TC)23 58 153~171 表 2 映山红种群4个不同年龄级的遗传多样性
Table 2. Genetic diversity of four different life stages of R. simsii
年龄级 Age group 样本数 Population size Na NE I HO HE h 幼苗 15 43 30.38 1.025 0 0.676 4 0.607 7 0.578 2 小树 15 50 36.52 1.188 5 0.881 2 0.690 7 0.654 4 成树 15 48 34.58 1.134 1 0.825 0 0.657 8 0.629 4 老树 15 47 35.77 1.133 7 0.846 3 0.672 8 0.632 6 注:Na:等位基因数;NE:有效等位基因数;I:Shannon多样性指数;HO:观察杂合度;HE:期望杂合度;h:Nei基因多样性指数。
Note: Na, NE, I, HO, HE, and h referred to number of alleles, effective number of alleles, Shannon's information index, observed heterozygosity, expected heterozygosity, and Nei's genetic diversity, respectively.表 3 映山红12个微卫星位点的遗传分化和基因流信息
Table 3. Summary of genetic variation and gene flow at 9 loci
位点 Locus Fis Fit Fst Nm HWE WRsE-37 -0.241 4 -0.156 9 0.068 1 3.419 3 0.000 0* WRsE-43 0.174 4 0.270 6 0.116 5 1.896 3 0.037 9 WRsE-70 -0.255 9 -0.169 7 0.068 6 3.393 4 0.001 9* WRsE-77 -0.073 8 -0.014 1 0.055 6 4.246 5 0.034 6 WRsE-81 -0.391 8 0.032 0 0.304 5 0.570 9 0.000 0* WRsE-82 -0.220 2 -0.104 5 0.094 8 2.387 3 0.000 2* WRsE-85 -0.408 3 -0.287 7 0.085 6 2.670 6 0.000 2* WRsE-90 -0.568 3 -0.534 3 0.021 7 11.271 0 0.001 1* WRsE-93 0.003 9 0.069 5 0.065 8 3.550 2 0.699 9 WRsE-97 -0.638 3 -0.615 1 0.014 2 17.404 7 0.000 0* WRsE-113 -0.561 3 -0.301 1 0.166 7 1.249 7 0.000 0* WRsE-131 -0.444 6 -0.247 4 0.136 5 1.581 6 0.007 1 平均 Mean -0.294 6 -0.162 1 0.102 4 2.192 4 注:*标注的是严重偏离Hardy-Weinberg平衡的位点。Fis:种群内近交系数;Fit:总近交系数;Fst:遗传分化系数;Nm:基因流、HWE:Hardy-Weinberg平衡
Note: * referring to locus significantly deviating from Hardy-Weinberg equilibrium. Fis, Fit, Fst, Nm, and HWE referred to intra-population inbreeding coefficient, total-population inbreeding coefficient, inter-population genetic differentiation coefficient (Fst), gene flow, and Hardy-Weinberg equilibrium, respectively -
种群内近交系数Fis变化范围为-0.638 3~0.174 4,平均值为-0.290 6;总近交系数Fit变化范围为-0.615 1~0.270 6,平均值为-0.162 1(表 3)。依据Nagylaki等[12]的理论,Fis和Fit平均值均为负数,表明映山红种群在检测的12个微卫星位点杂交现象严重,各种群以杂合子为主,尤其是WRsE-90、WRsE-97、WRsE-113位点。然而,在WRsE-43(Fis=0.174 4; Fit=0.270 6)和WRsE-93(Fis=0.003 9; Fit=0.069 5)位点存在着近交现象。
遗传分化系数Fst变化范围为0.014 2~0.304 5,平均值为0.102 4,表明仅10.24%的遗传变异发生在种群间,发生在种群内的遗传变异高达89.76%(表 3)。吕召云等[13]认为,Fst大于0.25时种群极度分化,Fst值在0.15和0.25之间种群间明显分化,Fst值在0.05和0.15之间种群间中度分化,Fst小于0.05种群间几乎无分化。在检测的12个位点处,WRsE-81位点极度分化,WRsE-113位点为明显分化,而WRsE-90和WRsE-97两个位点几乎无分化,其余8个位点为中度分化。
作为影响种群分化的重要因素,基因流Nm变化范围为0.570 9~17.404 7,平均值为2.192 4。WRsE-81(Nm=0.570 9)基因流水平非常低,种群在此位点很容易因遗传漂变而分化。其余11个微卫星位点的Nm值均大于1,足以抵抗种群遗传漂变导致的分化,尤其是杂交现象明显的位点WRsE-90(Nm=11.271 0)和WRsE-97 (Nm=17.404 7)。
-
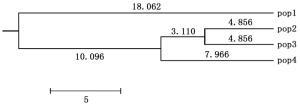
采用Nei氏方法计算4个世代的遗传距离(D)和遗传一致度(S),种群遗传一致度变化范围为0.583 1~0.867 6,遗传距离变化范围为0.142 0~0.563 9(表 4)。成树种群与小树种群的遗传一致度最大,其次是成树种群与老树种群之间,老树种群与幼苗种群的遗传一致度最小。相反,老树种群与幼苗种群之间的遗传距离最大,其次是成树与幼苗种群,遗传距离最小的是小树与成树种群。根据Nei's遗传距离,采用UPGMA方法构建聚类图,小树和成树种群先被聚在一起,然后与老树种群聚类在一个大分支,幼苗则独立被聚成一支(图 1)。
表 4 映山红种群不同年龄级间的Nei遗传一致度(右上)和遗传距离(左下)
Table 4. Genetic identify (above) and genetic distance (below) among different life stages of R. simsii population
幼苗
Seedlings小树
Juvenile成树
Adult老树
Old trees幼苗 Seedlings - 0.740 0 0.693 0 0.583 1 小树 Juvenile 0.301 2 - 0.867 6 0.766 7 成树 Adult 0.366 7 0.142 0 - 0.859 4 老树 Old trees 0.563 9 0.265 6 0.151 5 - -
植物遗传结构研究包括空间和时间2个层面,空间尺度上的遗传变异和时间尺度上的种群世代更替结合才能全面揭示植物种群的遗传多样性[14-15]。植物种群的遗传结构受到亲本瓶颈效应、遗传漂变、人为干扰等多种因素的综合影响,因此,分析种群遗传变异的时间动态,阐述种群不同阶段的遗传特征,探究种群不同世代遗传结构,能够准确反映种群整体遗传多样性程度,才能更好地预测种群的发展趋势[15-16]。Barker [17]认为,能够检测到4个以上等位基因的微卫星标记能够较好地分析种群遗传多样性。本研究选用的12个标记检测的等位基因数为3~9个,平均5.08个,能够反映映山红种群遗传多样性特征。
映山红生命周期长,多数野生个体年龄在数百年以上,通过追踪种群世代过程研究种群遗传结构的时间动态变化非常困难,因此,采用不同年龄级个体之间的遗传差异进行研究,即“空间代时间”的年龄级法进行研究[14],本研究所选的4个年龄级可以代表映山红的4个生活史阶段。平均期望杂合度反映遗传多样性的程度,并且不受取样大小的影响[18]。Takezaki等[19]认为,微卫星位点的期望杂合度在0.3~0.8的范围内则具有较高的遗传多样性。本研究选取的4个种群期望杂合度为0.607 7~0.690 7,Shannon信息指数为1.025 0~1.188 5,表明大别山黄狮寨野生映山红种群遗传多样性非常丰富,明显高于冷杉(HE=0.234 4,I= 0.376 4)、银杏(HE=0.250 8,I=0.359 9)、水松(HE=0.163 1;I=0.247 7)等乔木,但却低于菊科植物Erigeron arisolius(HE=0.748)[15, 20-22]。较高的种群遗传多样性同样存在于井冈山杜鹃种群(HE= 0.642)和大白杜鹃种群(HE=0.758),本研究进一步证实了野生多年生植物具有较高的遗传多样性的结论[10, 23]。
近交系数Fis和Fit值在-1~1范围内,大于0表示杂合子缺失,小于0则为杂合子过剩[24]。本研究检测的12个微卫星位点,10个位点的近交系数均为负值,表明该映山红种群杂合子显著过剩,近交衰退对映山红遗传分化的影响不明显。检测的12个位点遗传分化程度差异显著,但大多数属于中度分化(0.05 < Fst < 0.15)。各种群间基因交流水平高时(Nm>1),种群能够抵抗遗传漂变造成的分化;基因流水平低时(Nm < 1),种群很可能因为遗传漂变而产生分化[25]。本研究检测的12位点处,仅1个位点处的Nm小于1,如此频率的基因流动能够抑制遗传漂变导致的种群分化。在大树杜鹃中,基因流也比较频繁,平均Nm为4.065 [3]。
4个龄级的映山红种群遗传多样性大小为:小树种群>成树种群>老树种群>幼苗种群,表明映山红从幼苗成长到小树阶段,遗传多样性递增,与水松和厚壳桂等乔木的研究结果一致[15, 26]。然而,映山红从小树阶段逐渐发育到老树阶段,种群遗传变异有衰退趋势,此现象与长叶榧种群中的观察结果一致[14]。Nei’s遗传距离越小,亲缘关系越近;遗传一致度越小,亲缘关系越远[27]。小树与成树的遗传一致度最大,而老树与幼苗的遗传一致度最小。分子方差分析表明,映山红不同年龄级种群的遗传变异主要来自于种群内,仅10.24%的遗传变异发生在不同年龄级种群间,高于井冈山杜鹃花种群(4.60%)[10],低于大树杜鹃种群(22.0%)[3]。
不同年龄级个体间遗传分化的差异,可能是不同年份交配格局造成的,繁殖个体自身的变化以及自然选择同样对龄级间的遗传分化造成了影响。另外,映山红花期短,荚果种子数量虽多但是扩散能力差,而且种子在野外萌发困难,幼苗不易成活,野外考察发现,林下的实生苗极少,因此,有效种群规模难以扩大。野外考察还发现,映山红的幼苗大多生活在成树和老树的底层,对水分、光照等资源竞争能力相对较弱。老树丛枝较多,主枝相对于小树和成树而言,生长较慢。在该映山红种群中,小树的生命力旺盛,更容易发生遗传变异而积累丰富的遗传多样性。为开发生态旅游而引起的生境地片段化及人工干扰使得映山红个体分布不连续,成树和小树基本上来源于几株老树母株,因此,遗传相似度高,遗传距离小。本研究丰富了目前杜鹃花科物种进化的研究案例,也为大别山野生杜鹃资源的保护提供了科学依据。然而,引起大别山映山红种群分化的原因较多,需进一步深入研究。
-
不同龄级的映山红遗传多样性差别较大,幼苗和老树种群多样性最差,小树种群多样性最丰富,且各龄级种群内主要繁殖方式为杂交。映山红种群89.76%的遗传变异存在于年龄级内,仅10.24%存在于年龄级间。不同龄级间的基因流水平高,12个位点中仅有1个位点Nm值小于1。4个龄级中,遗传一致度最高的为小树和成树种群
大别山不同龄级映山红种群遗传多样性的SSR分析
Genetic Diversity of Rhododendron simsii Populations on Dabieshan at Different Life Stages Based on SSR Markers
-
摘要:
目的 利用SSR标记比较大别山黄狮寨不同年龄级映山红(Rhododendron simsii Planch.)种群的遗传多样性及遗传结构,探究映山红不同世代间遗传多样性的变化规律,为大别山野生映山红资源的合理利用和高效保护提供科学依据。 方法 按照基径大小和丛枝多少将大别山黄狮寨典型映山红种群划分为老树、成树、小树、幼苗4个年龄级,筛选出12对多态性强的SSR引物用于PCR扩增,扩增产物经6%变性聚丙烯酰胺凝胶电泳检测并银染。构建"0/1"矩阵,利用POPGENE 32.0软件分析种群遗传多样性。基于Nei's遗传距离,采用软件MEGA5.0进行UPGMA聚类。 结果 不同龄级的映山红遗传多样性差别较大,幼苗和老树种群多样性最差,小树种群多样性最丰富。12个微卫星标记观测等位基因数为3~9个,平均5.08;有效等位基因数为2.254 9~6.129 7,平均3.460 5;观察杂合度HO和期望杂合度HE分别为0.676 4~0.881 2和0.607 7~0.690 7。Shannon信息指数(I)以小树群体最高,成树次之,幼苗最低。近交系数Fis为-0.638 3~0.174 4,平均为-0.294 6;总近交系数Fit为-0.615 1~0.270 6,平均为-0.162 1,表明各龄级种群内主要繁殖方式为杂交。分子方差分析(AMOVA)表明89.76%的遗传变异存在于年龄级内,仅10.24%存在于年龄级间。基因流水平高,仅1个位点Nm < 1。遗传一致度最高的为小树和成树种群。 结论 大别山黄狮寨映山红种群遗传多样性丰富,种群间中度分化,遗传变异主要存在于年龄级内。 Abstract:Objective Microsatellite markers were used to compare the genetic diversity and genetic structure of Rhododendron simsii populations at four different life stages sampled from Huangshizhai of Dabieshan region to clarify the mechanism of genetic diversity among different age groups. Method The populations of R. simsii were divided into four age groups (old trees, adult, juvenile, and seedlings) according to their sizes of basal diameter and numbers of branches. Twelve SSR markers were used for PCR amplification, and the products were subjected to 6% polyacrylamide gel electrophoresis, which were further visualized through silver staining. The "0/1" matrix was constructed and POPGENE 32.0 software was used to analyze the genetic diversity. Based on Nei's genetic distance, UPGMA dendrogram was constructed with MEGA 5.0 software. Result Different genetic diversities were observed in the four R. simsii populations, the lowest genetic diversity was observed in seedlings population, followed by old trees population, while the highest genetic diversity was observed in juvenile population. The observed and effective number of alleles per locus ranged from 3 to 9 and 2.254 9-6.129 7, with the average of 5.08 and 3.460 5, respectively. Moreover, HO and HE were in the ranges of 0.676 4-0.881 2 and 0.607 7-0.690 7, respectively. The Shannon's information index (I) was the highest in juvenile population, followed by adult population, and lowest in seedling population. Fis and Fit ranged within-0.638 3-0.174 4 and-0.615 1-0.270 6, with the mean of-0.294 6 and-0.162 1, indicating the outcross took place frequently. In particular, the AMOVA analysis showed that 89.76% of genetic variation was maintained within age groups, while only 10.24% presented among age groups. Frequent gene flow was observed, as only one locus showed Nm less than 1.0.The genetic identify between adult and juvenile groups was the highest. Conclusion High genetic diversity existed among R. simsii populations located on Huangshizhai. Moderate differentiation was observed, which was maintained between age groups. -
Key words:
- Rhododendron simsii Planch.
- / life stage
- / genetic diversity
- / population structure
- / microsatellite
-
表 1 微卫星标记信息
Table 1. Information of microsatellite markers
位点 Locus 正向引物(5′-3′)
Forward primer (5′-3′)反向引物(5′-3′)
Reverse primer (5′-3′)基序
Motif退火温度 Annealing temperature /℃ 片段大小
Size range /bpWRsE-37 CTTGCCACTTTGAGTTTGAG AGAGTAATTTGGAGGAAGCG (CT)28 55 200~212 WRsE-43 TGAACCCTTCTTCTTCTTCC TTTGATTGAAGGGTGGAGTG (TC)23 56 249~262 WRsE-70 TCTTCCGATTCCATCATTCC TGGGCGTGATTTGGTTATAA (CT)22 58 178~192 WRsE-77 TAGCTGCTTACTGTTGAAGG AGTAAAAGGGCTGAAACTGT (AG)22 57 149~165 WRsE-81 GCCCTATCCCTCAACTTTAC GAGGAGCGTGGTTAGTAATT (TC)21 56 229~253 WRsE-82 GTATGGGACCTGTGATTTCC CTCCAACTAGCTACTCCAAC (GA)24 57 228~236 WRsE-85 GAAATCTCGAATCACCTCCA AAGGTGTTGGTGGACTAATC (TC)21 56 149~167 WRsE-90 TTGAAGAACACTCAAGTTGC ACGTAGAACATTGCTTTCCT (GA)21 57 196~202 WRsE-93 GGTATCCGGTTTTCATCACT ATACCCACTAGCAACAGAGA (GA)23 57 233~256 WRsE-97 AGAAAACTGGGAGATGTGTC AGGTGATCATCTTTGCATGT (CT)21 58 222~246 WRsE-113 TATTGTACAGCTCCCCTTTG CCTCAATGTTCTATCGACGT (CT)23 57 185~207 WRsE-131 CTCTTTCCCCTTTCATGTGA AAGTGTTGAGTCCTCGTATG (TC)23 58 153~171 表 2 映山红种群4个不同年龄级的遗传多样性
Table 2. Genetic diversity of four different life stages of R. simsii
年龄级 Age group 样本数 Population size Na NE I HO HE h 幼苗 15 43 30.38 1.025 0 0.676 4 0.607 7 0.578 2 小树 15 50 36.52 1.188 5 0.881 2 0.690 7 0.654 4 成树 15 48 34.58 1.134 1 0.825 0 0.657 8 0.629 4 老树 15 47 35.77 1.133 7 0.846 3 0.672 8 0.632 6 注:Na:等位基因数;NE:有效等位基因数;I:Shannon多样性指数;HO:观察杂合度;HE:期望杂合度;h:Nei基因多样性指数。
Note: Na, NE, I, HO, HE, and h referred to number of alleles, effective number of alleles, Shannon's information index, observed heterozygosity, expected heterozygosity, and Nei's genetic diversity, respectively.表 3 映山红12个微卫星位点的遗传分化和基因流信息
Table 3. Summary of genetic variation and gene flow at 9 loci
位点 Locus Fis Fit Fst Nm HWE WRsE-37 -0.241 4 -0.156 9 0.068 1 3.419 3 0.000 0* WRsE-43 0.174 4 0.270 6 0.116 5 1.896 3 0.037 9 WRsE-70 -0.255 9 -0.169 7 0.068 6 3.393 4 0.001 9* WRsE-77 -0.073 8 -0.014 1 0.055 6 4.246 5 0.034 6 WRsE-81 -0.391 8 0.032 0 0.304 5 0.570 9 0.000 0* WRsE-82 -0.220 2 -0.104 5 0.094 8 2.387 3 0.000 2* WRsE-85 -0.408 3 -0.287 7 0.085 6 2.670 6 0.000 2* WRsE-90 -0.568 3 -0.534 3 0.021 7 11.271 0 0.001 1* WRsE-93 0.003 9 0.069 5 0.065 8 3.550 2 0.699 9 WRsE-97 -0.638 3 -0.615 1 0.014 2 17.404 7 0.000 0* WRsE-113 -0.561 3 -0.301 1 0.166 7 1.249 7 0.000 0* WRsE-131 -0.444 6 -0.247 4 0.136 5 1.581 6 0.007 1 平均 Mean -0.294 6 -0.162 1 0.102 4 2.192 4 注:*标注的是严重偏离Hardy-Weinberg平衡的位点。Fis:种群内近交系数;Fit:总近交系数;Fst:遗传分化系数;Nm:基因流、HWE:Hardy-Weinberg平衡
Note: * referring to locus significantly deviating from Hardy-Weinberg equilibrium. Fis, Fit, Fst, Nm, and HWE referred to intra-population inbreeding coefficient, total-population inbreeding coefficient, inter-population genetic differentiation coefficient (Fst), gene flow, and Hardy-Weinberg equilibrium, respectively表 4 映山红种群不同年龄级间的Nei遗传一致度(右上)和遗传距离(左下)
Table 4. Genetic identify (above) and genetic distance (below) among different life stages of R. simsii population
幼苗
Seedlings小树
Juvenile成树
Adult老树
Old trees幼苗 Seedlings - 0.740 0 0.693 0 0.583 1 小树 Juvenile 0.301 2 - 0.867 6 0.766 7 成树 Adult 0.366 7 0.142 0 - 0.859 4 老树 Old trees 0.563 9 0.265 6 0.151 5 - -
[1] 冯国楣.花中西施杜鹃花[J].园林, 1998(12):124-125. [2] Wang S, Li Z, Jin W, et al. Development and characterization of polymorphic microsatellite markers in Rhododendron imsii (Ericaceae)[J]. Plant Species Biology, 2017, 32(1):100-103. doi: 10.1111/psbi.2017.32.issue-1 [3] Wu F Q, Shen S K, Zhang X J, et al. Genetic diversity and population structure of an extremely endangered species:the world's largest rhododendron[J]. AoB Plants, 2014, 7(22):10696-10700. [4] Liu Y F, Xing M, Zhao W, et al. Genetic diversity analysis of Rhododendron aureumgeorgi (Ericaceae) located on Changbai mountain using ISSR and RAPD markers[J]. Plant Systematics and Evolution, 2012, 298(5):921-930. doi: 10.1007/s00606-012-0601-0 [5] Kuttapetty M, Pillai P P, Varghese R J, et al. Genetic diversity analysis in disjunct populations of Rhododendron arboreum from the temperate and tropical forests of Indian subcontinent corroborate Satpura hypothesis of species migration[J]. Biologia, 2014, 69(3), 311-322. [6] Zhao B, Xu M, Si G C, et al. Genetic diversity and genetic differentiation of Rhododendron concinnum wild populations in Qinling mountains of northwest China:an AFLP analysis[J]. Chinese Journal of Applied Ecology, 2012, 23(11):2983-2990. [7] Wang S, Pan L, Hu K, et al. Development and characterization of polymorphic microsatellite markers in Momordica charantia (Cucurbitaceae)[J]. American Journal of Botany, 2010, 97(8):75-78. doi: 10.3732/ajb.1000153 [8] 李久林, 梁士楚, 廖凤林.马缨杜鹃种群密度的GM(1, 1)模型[J].贵州师范大学学报:自然科学版, 1998(2):15-18. [9] Wu Z H, Wang S Z, Hu J H, et al. Development and characterization of microsatellite markers for Sagittariatrifolia var. sinensis (Alismataceae)[J]. American Journal of Botany, 2011, 98(2):36-38. doi: 10.3732/ajb.1000434 [10] Li M, Chen S, Shi S, et al. High genetic diversity and weak population structure of Rhododendron jinggangshanicum, a threatened endemic species in mount Jinggangshan of China[J]. Biochemical Systematics & Ecology, 2015, 58:178-186. [11] Hu J, Pan L, Liu H, et al. Comparative analysis of genetic diversity in sacred lotus (Nelumbonucifera gaertn.) using AFLP and SSR markers[J]. Molecular Biology Reports, 2012, 39(4):3637-3647. doi: 10.1007/s11033-011-1138-y [12] Nagylaki T. Fixation indices in subdivided populations[J]. Genetics, 1998, 148(3):1325-1332. [13] 吕召云, 杨茂发, 师沛琼, 等.贵州省烟蚜遗传多样性分析[J].中国农业科学, 2013, 46(13):2685-2694. doi: 10.3864/j.issn.0578-1752.2013.13.007 [14] 李建辉, 金则新, 李钧敏.濒危植物长叶榧群体不同年龄级遗传多样性的RAPD和ISSR分析[J].浙江大学学报:理学版, 2010, 37(1):104-109. [15] 吴则焰, 刘金福, 洪伟, 等.孑遗植物水松不同年龄级种群遗传多样性的ISSR分析[J].生态学杂志, 2012, 31(8):1911-1916. [16] Su Y J, Wang T, Ouyang P Y. High genetic differentiation and variation as revealed by issr marker in Pseudotaxus chienii (Taxaceae), an old rare conifer endemic to China[J]. Biochemical Systematics & Ecology, 2009, 37(5):579-588. [17] Barker J S F. A global protocol for determining genetic distances among domestic livestock breeds: proceedings of the 5th world congress on genetics applied to livestock production[D]. Canada: University of Guelph, 1994. [18] Frankham R, Ballou J D, Briscoe D A. Introduction to Conservation Genetics[M]. New York:Cambridge University Press, 2002. [19] Takezaki N, Nei M. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA[J]. Genetics, 1996, 144(1):389-399. [20] 王燕, 唐绍清, 李先琨.濒危植物元宝山冷杉的遗传多样性研究[J].生物多样性, 2004, 12(2):269-273. doi: 10.3321/j.issn:1005-0094.2004.02.007 [21] Tian S, Luo L C, Song G E, et al. Clear genetic structure of Pinus kwangtungensis (Pinaceae) revealed by a plastid DNA fragment with a novel minisatellite[J]. Annals of Botany, 2008, 102(1):69-78. doi: 10.1093/aob/mcn068 [22] Edwards C E, Lindsay D L, Bailey P, et al. Patterns of genetic diversity in the rare Erigeron lemmoniand comparison with its more widespread congener, Erigeron arisolius (Asteraceae)[J]. Conservation Genetics, 2014, 15(2):419-428. doi: 10.1007/s10592-013-0549-9 [23] Wang X Q, Huang Y, Long C L. Assessing the genetic consequences of flower-harvesting in Rhododendron decorum Franchet (Ericaceae) using microsatellite markers[J]. Biochemical Systematics & Ecology, 2013, 50(10):296-303. [24] Klocke J A, Hu M Y, Chiu S F, et al. Grayanoidditerpene insect antifeedants and insecticides from Rhododendron molle[J]. Phytochemistry, 1991, 30(6):1797-1800. doi: 10.1016/0031-9422(91)85015-R [25] Whitlock M C, Mccauley D E. Indirect measures of gene flow and migration:FST≠1/(4Nm+1)[J]. Heredity, 1999, 82(2):117-125. doi: 10.1038/sj.hdy.6884960 [26] 高三红, 王峥峰, 张军丽, 等.鼎湖山厚壳桂(Cryptocarya chinensis)不同年龄级种群遗传多样性[J].中山大学学报:自然科学版, 2005, 44(s1):209-212. [27] 师沛琼, 杨茂发, 吕召云, 等.贵州省烟青虫遗传多样[J].中国农业科学, 2014, 47(9):1836-1846. doi: 10.3864/j.issn.0578-1752.2014.09.019 -




 下载:
下载: