-
血皮槭(Acer griseum(Franch.)Pax)为中国特有种,具有极高的观赏价值。1993年,血皮槭获得英国皇家园艺学会园艺奖,成为国内外著名的景观树种[1]。除观赏价值外,血皮槭的许多价值尚待开发和利用,例如木材坚硬,可作为良好的建筑材料;木材纹理精美可制作乐器及工艺品。但是,这个极具开发价值的树种正濒临灭绝,急需保护。血皮槭的分布范围仅限于我国中部地区海拔800~2 000 m的山地,分布区严重片段化,天然群体数量非常小,表现出濒危物种的典型特征[2]。根据世界自然保护联盟(IUCN)的标准,血皮槭在《中国物种红色目录》中已被列为濒危树种(EN A2c)[3]。
植物叶绿体基因组DNA(Chloroplast DNA,cpDNA)的研究为珍稀濒危物种制定合理有效的保护策略提供了重要的理论依据。通常,叶绿体非编码区的碱基替换率较高,为中性突变,不受自然选择等其它因素的影响,是研究植物谱系地理、分子系统发育和群体遗传学重要的工具,近年来被成功用于种间或种内变异水平的研究[4]。例如,利用cpDNA序列对濒危植物南方红豆杉(Taxus wallichiana var. mairei (Lemée& H.Léveillé) L. K. Fu & Nan Li),日本红枫(Acer pycnanthum K. Koch),瘿椒树(Tapiscia sinensis Oliv.),青钱柳(Cyclocarya paliurus (Batal.) Iljinsk.),珙桐(Davidia involucrata Baill.),金钱槭(Dipteronia sinensis Oliv.)等植物[5-10]开展了谱系地理学研究。近年来,对于濒危植物血皮槭的研究主要集中在SSR引物开发、天然群体遗传变异和谱系地理学初步研究等方面[11-13],其中王佳慧利用rps16序列对血皮槭谱系地理学进行初步探讨取得进展。但是一个序列并不能全面反映血皮槭的谱系地理结构,本研究利用20个通用cpDNA序列,以血皮槭16个天然群体的16株个体为材料,筛选适宜于血皮槭种内研究的高变异率cpDNA非编码区序列,为进一步研究濒危种血皮槭的分子谱系地理学奠定基础,对于制定血皮槭合理的保护策略具有重要意义。
HTML
-
试验材料来源于本课题组野外调查的16个血皮槭天然群体(表 1),每个群体随机选取1株个体为试材。新鲜叶片经硅胶充分干燥后,置于-80℃冰箱保存备用。
群体
Population代码
Code纬度/N
Latitude经度/E
Longitude海拔/m
Altitude群体
Population代码
Code纬度/N
Latitude经度/E
Longitude海拔/m
Altitude重庆巫溪 WX 31°28'17" 109°50'59" 1280 湖北兴山 XS 31°19'18" 110°29'08" 1 450 重庆城口 CHK 32°08'20" 108°34'45" 1127 湖北五峰 WF 30°10'20" 110°35'20" 1 496 陕西太白山 TBS 34°04'49" 107°42'48" 1668 湖北神农架 SNJ 31°27'41" 110°23'26" 1 272 陕西华山 HS 34°28'15" 110°04'14" 1989 河南西峡 TPZ 33°39'27" 110°37'23" 837 山西阳城 YC 35°16'18" 112°26'28" 800 河南嵩县 SX 34°13'60" 111°50'48" 941 湖南张家界 ZJJ 29°03'09" 110°28'38" 1440 河南内乡 NX 33°29'38" 111°55'42" 1 234 湖南沅陵 YL 28°53'39" 110°24'46" 561 河南栾川 LC 33°44'28" 111°37'50" 1 031 湖南壶瓶山 HPS 30°02'20" 110°31'23" 1640 甘肃天水 TS 34°14'10" 105°58'33" 1 390 Table 1. The geographical location of the sampled populations of A. griseum
-
取干燥叶片30 mg,采用DNA提取试剂盒DP305 (Tiangen Biotech Co., China) 提取血皮槭叶片总DNA。采用0.8%的琼脂糖凝胶进行电泳,G-box凝胶成像仪(Chemi XX6,Syngene)拍照检测DNA的质量,酶标仪(Spectra Max i3)检测DNA纯度和浓度。DNA浓度稀释到50 ng·μL-1,放于-20℃冰箱保存备用。
-
从参考文献[14-17]挑选了20对适合种内水平的cpDNA非编码通用引物,由上海美吉生物医药科技有限公司合成(表 2)。为对20对通用引物进行初步筛选,随机挑选2个样品进行PCR反应。反应体系20 μL,包括模板DNA 50 ng、2×Buffer Master Mix 10 μL、10 μmol·L-1 F-primer 0.4 μL、10 μmol·L-1 R-primer 0.4 μL、其余ddH2O补齐。NdhF-rpl32,psbJ-petA序列的PCR程序:80℃预变性5 min;95℃变性1 min,50℃退火1 min,0.3℃·s-1升温到65℃,65℃延伸4 min,30个循环;65℃延伸5 min。其他序列的PCR程序:94℃预变性5 min;94℃变性45 s,55℃退火45 s,72℃延伸45 s,30个循环;72℃延伸10 min。PCR产物采用1%琼脂糖凝胶检测,筛选出条带清晰单一的引物;然后利用初筛合格的引物对所有样品进行PCR扩增,选择条带清晰明亮、无非特异性扩增的PCR扩增产物,送到上海美吉生物医药科技有限公司纯化,然后采用ABI 3730xl DNA analyzer进行测序。
编号
Code序列名称
Sequence name引物信息(5’-3’)
Primer details编号
Code序列名称
Sequence name引物信息(5’-3’)
Primer details1 trnH-psbA F: CGCGCATGGTGGATTCACAATCC
R: GTTATGCATGAACGTAATGCTC11 rpl16 F: GCTATGCTTAGTGTGTGACTCGTTG
R: CCCTTCATTCTTCCTCTATGTTG2 psbJ-petA F: ATAGGTACTGTARCYGGTATT
R: AACARTTYGARAAGGTTCAATT12 rpl32-trnL F: CTGCTTCCTAAGAGCAGCGT
R: CAGTTCCAA AA A AACGTACTTC3 ndhF-rpl32 F: CCAATATCCCTT YYTTTTCCAA
R: GAAAGGTATKATCCAYGMATATT13 rpoB-trnC F: CACCCRGATTYGAACTGGGG
R: CKACAA AAYCCYTCRAATTG4 trnD-trnT F: ACCAATTGAACTACAATCCC
R: CTACCACTGAGTTAAAAGGG14 rps16-trnK F: AAAGTGGGTTTTTATGATCC
R: TTAAAAGCCGAGTACTCTACC5 atpB-rbcL F: GTGGAAACCCCGGGACGAGAAGTAGT
R: ACTTGCTTTAGTTTCTGTTTGTGGTGA15 rpl20-rps18 F: CTTCGTCGTTTGTGGATTAC
R: AGTCGATTTATTAGTGAGCA6 trnS-trnG F: AGATAGGGATTCGAACCCTCGGT
R: GTAGCGGGAATCGAACCCGCATC16 psbD-trnT F: CTCCGTARCCAGTCATCCATA
R: CCCTTTTAACTCAGTGGTAG7 ndhF F: GATTTGATTTTCCCCTTATC
R: TCCATAGCATCAGGTAACC17 psbM-trnD F: AGCAATAAATGCRAGAATATTTACTTCCAT
R: GGGATTGTAGYTCAATTGGT8 trnL-trnF F: CGAAATCGGTAGACGCTACG
R: ATTTGAACTGGTGACACGAG18 trnV-ndhC F: GTCTACGGTTCGARTCCGTA
R: TATTATTAGAAATGYCCARAA AATATCATATTC9 petB-petD F: CTATCGTCCRACCGTTACWGAGGCT
R: CAAAYGGATAYGCAGGTTCACC19 psbB-psbH F: TCCAAAAANKKGGAGATCCAAC
R: TCAAYRGTYTGTGTAGCCAT10 trnS-trnfM F: GAGAGAGAGGGATTCGAACC
R: CATAACCTTGAGGTCACGGG20 rps12-rpl20 F: ATTAGAAANRCAAGACAGCCAAT
R: CGYYAYCGAGCTATATATCCTable 2. Twenty pairs of the cpDNA universal primer sequence
-
根据原始峰图,使用Contig Express软件(Informax Inc.,North Bethesda,MD,USA)对cpDNA测序结果进行峰图检查及拼接,手工校对。然后,采用BioEdit v7.0.5.2软件[18]分别对cpDNA序列进行比对和手工调整;利用MEGA v5.0[19]计算信息位点。cpDNA序列的变异位点,都是潜在的信息位点(PICs),包括核苷酸的替换、倒置、插入和缺失[11]。利用MEGA v5.0进行聚类分析,采用邻接法(Neighbor joining)构建系统发育树,自展检验(bootstrap)通过1 000次重复获得。为获得血皮槭潜在信息位点(PICs)和变异率(V),引入公式:
式(1)、(2)中:I表示序列插入和缺失数,S表示序列核苷酸替换数,R表示序列倒置数,A表示序列比对后长度。
1.1. 试验材料
1.2. 方法
1.2.1. 基因组DNA提取与检测
1.2.2. cpDNA通用引物筛选及测序
1.2.3. cpDNA序列编辑和分析
-
酶标仪检测提取的DNA纯度和浓度都较高。OD260 /OD280为1.606~1.992,DNA浓度为42.8~154.2 ng·μL-1,符合PCR反应所需的要求。电泳结果显示DNA条带清晰,满足后续试验要求。
-
所有20对cpDNA通用引物都能扩增出目的片段。其中9个cpDNA序列(表 2编号1-9)检测到不同程度的变异位点(表 3),序列(表 2编号5-9)只检测到一个位点的变异(数据省略)。其余11个cpDNA序列(表 2编号10-20)未能检测到变异位点。
序列Sequence psbJ-petA ndhF-rpl32 trnD-trnT trnH-psbA 群体名称
Population name2
1
94
9
75
4
34
1
96
2
67
3
67
3
74
5
25
3
92
8
92
9
02
9
3重庆巫溪(WX) C T - G - A A - T - - - 重庆城口(CHK) A - - G - A - - T A - ☆ 陕西太白山(TBS) A - - G - A - - T A - - 陕西华山(HS) A - - G - A - - C A - - 山西阳城(YC) A - - G - A - - C A - - 湖南张家界(ZJJ) A - - G △ A - - T - - - 湖南沅陵(YL) A - - G △ A - - T A - - 湖南壶瓶山(HPS) A - - G △ A - - T A - - 湖北兴山(XS) A - - G - A - - T A A - 湖北五峰(WF) A - - G △ A - - T A - - 湖北神农架(SNJ) C T - G - A A - T A - - 河南西峡(TPZ) A - - G - A - - T - - - 河南嵩县(SX) A - A G - A - - T A - - 河南内乡(NX) A - - C - A - ◇ C A - - 河南栾川(LC) A - A G - A - - C A A - 甘肃天水(TS) A - - G - - - - T A - - 测序长度(bp) 867 876 620 450 比对长度(bp) 563 746 574 395 注:“△”:TAACTCCGAAATACAAAAAAGAGAGAGAGATAGAAAGGTATC ACAAA;“◇”:TTCTTTCACT;“☆”:TAATAAA;“-”:缺失
Note: “△”:TAACTCCGAAATACAAAAAAGAGAGAGAGATAGAAAGGTA TCACAAA;“◇”:TTCTTTCACT;“☆”:TAATAAA;“-”:deletion.Table 3. Variation sites of 4 cpDNA sequences in the populations of A. griseum
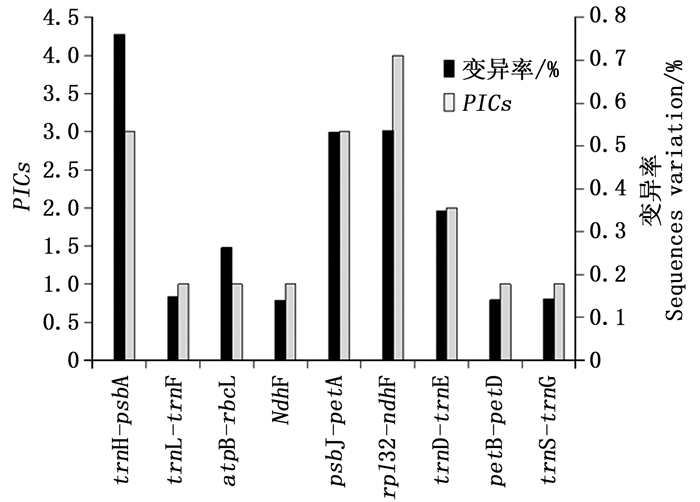
对9个序列的PICs值和变异率(图 1)进行比较,筛选出4个变异丰富的cpDNA序列,按照PICs值由高到低依次为ndhF-rpl32,psbJ-petA,trnH-psbA,trnD-trnT,变异率由高到低依次为trnH-psbA,ndhF-rpl32,psbJ-petA,trnD-trnT。
血皮槭cpDNA非编码区序列的种内变异(PICs值)与其他木本植物相比很低(见表 4)。rps12-rpl20和rpl32-trnL序列在血皮槭中表现无变异,而在伞花木[20](Eurycorymbus cavaleriei(Le′vl.) Rehd. et Hand.-Mazz.)群体和珙桐[9](D. involucrata)群体检测到不同程度的变异;trnH-psbA,atpB-rbcL,ndhF-rpl32和psbJ-petA序列在血皮槭中的PICs值与其他木本植物如色木槭[21](Acer mono Maxim.)、红枫[22](Acer rubrum L.)、美洲糖槭[23](Acer saccharum Marsh.)和观光木[24](Michelia odora)相比很低。
序列
sequencePICs值PICs value 血皮槭
A. griseum伞花木
E. cavaleriei珙桐
D. involucrata色木槭
A. mono观光木
M.odora日本红枫
A.rubrum美洲糖槭
A.saccharumrps12-rpl20 0(0) 2(1) - - - - - rpl32-trnL 0(0) - 13(11) - - - - trnH-psbA 3(0) - - 20(14) 5(4) - - atpB-rbcL 1(1) - - - - 8(2) - ndhF-rpl32 4(1) - - - - - 27(15) psbJ-petA 3(1) - - - - - 20(12) 群体数量 16 18 24 63 21 115 16 个体数量 16 170 231 1236 161 258 233 注:括号内的数字为核苷酸替换数
Note:The number in parentheses represents the number of nucleotide substitutionsTable 4. PICs values comparison of different cpDNA sequences in different tree species
-
基于4个序列ndhF-rpl32,psbJ-petA,trnH-psbA和trnD-trnT的比对结果,构建了血皮槭群体的系统发育树(图 2)。此系统发育树显示,16个群体明显分为两大支,SNJ、WX、ZJJ、TPZ、TS、TBS、YL、WF、CHK、HPS、SX、XS群体聚为一支,LC、YC、HS、NX群体聚为一支。SNJ、WX群体和NX群体分别从两大支单独分离出来。位于大巴山脉东部的重庆巫溪(WX)和湖北神农架(SNJ)同聚为一支,位于秦岭山脉东部的河南栾川(LC)和内乡(NX)、山西阳城(YC)和陕西华山(HS)群体聚为一支。
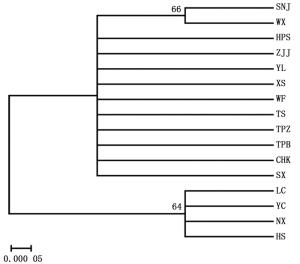
Figure 2. NJ phylogenetic tree based on 4 cpDNA seqences of A. griseum(population codes are shown in Table 1)
2.1. DNA提取与PCR扩增
2.2. 序列分析
2.3. 系统发育树构建
-
与其他一些木本植物相比,血皮槭种内变异率较低。血皮槭的种内变异率较低的原因可能与只选用了较少的16个样品用于cpDNA序列筛选有关,在植物的谱系地理学研究中,天然群体的数量非常重要,样本数量越多越能全面反映其遗传变异情况;也可能与血皮槭及天然群体分布范围窄且呈片段化分布的特点有关,一般来说,广域分布的物种比狭域分布的物种拥有更丰富的遗传变异。例如广域分布的槭属植物如色木槭和糖槭的cpDNA序列变异率比狭域分布的日本红枫和血皮槭高很多。
通过构建系统发育树分析,16个群体明显分为两大支,而且两个分支内群体间地理距离较近,推测这两个分支的群体经历了长期的地理隔离,与其它群体之间缺乏有效的基因流。ZJJ、TPZ、TS、TBS、YL、WF、CHK、HPS、SX和XS群体位于秦岭山脉、大巴山脉和武陵山脉,此区域位于华中地区的中心,独特的地形和复杂的气候是众多动植物的避难所,为物种的分化和地理隔离提供先决条件。此外,叶绿体基因流是通过种子进行母系传播,基于血皮槭翅果种子传播距离有限、空种率高和种子难以萌发等特性,加之恶劣生境的影响,限制了种子的传播,阻断了基因流,从而使血皮槭在区域内分化和地理隔离成为可能。
-
本研究通过对20个血皮槭叶绿体DNA非编码区差异序列进行筛选,共筛选出4个多态性较好的序列(psbJ-petA、ndhF-rpl32、trnD-trnT和trnH-psbA)。利用筛选得到的4个cpDNA序列对血皮槭16个群体进行系统发育分析,结果显示16个群体应分为两大类。本研究筛选获得的4个cpDNA序列在血皮槭种群内拥有相对丰富的变异,是研究种内变异良好的分子标记,可以进一步用于血皮槭系统发育、遗传变异和谱系地理学研究。




 DownLoad:
DownLoad:
