-
竹亚科牡竹属的巨龙竹(Dendrocalamus sinicus Chia et J. L. Sun)为我国云南特有大型丛生竹种,其胸径可达30 cm,秆高可达30 m以上,是目前已知世界上最高大粗壮的竹种[1]。巨龙竹秆形在自然分布区中存在两种遗传稳定的变型,即“通直型”和“弯曲型”[2]。弯曲型竹秆基部常畸变,呈短缩、鼓胀、歪扭等形状,不利于竹板材加工;通直型巨龙竹秆形通直,平均单株质量100150 kg,单位面积年均产材量比我国主要经济竹种毛竹(Phyllostachys edulis(Carr.) H. de Lehaie.)高58倍[3]。通直型巨龙竹是培育特大型工业用材的优异竹种之一,也是我国南部热区竹产业发展和生态环境建设中最具发展潜力的优良经济竹种之一[3-4]。秆形作为巨龙竹资源培育与利用的关键性状之一,其发育机制尚不清楚,这给巨龙竹优良种源的选择和培育带来了障碍。同时,“通直型”和“弯曲型”巨龙竹作为一对自然的变异类型,也是木本竹种秆形发育研究中理想的对照系。目前,作者正在开展巨龙竹秆形发育的转录组分析,试图解析不同秆形发育过程中关键基因的时空表达模式。
实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)技术因其特异性强、灵敏度高、快速准确、重复性好等[5-6]优点被广泛用于基因表达分析。在植物学研究中,常选用稳定表达的内参基因转录延伸因子-1α(EF-1α)、甘油醛-3-磷酸-脱氢酶(GAPDH)、肌动蛋白基因(Actin)、微管蛋白基因(Tubilin)为内参基因[7-8]。随着基因芯片和基因组学的发展,又挖掘了新的内参基因蛋白磷酸酶2A(PP2A)和液泡膜内嵌蛋白基因(TIP-41)等[8]。各内参基因在不同组织、不同发育时期、不同处理中的稳定性也存在差异[8]。近期有关竹亚科植物内参基因的筛选已有一些相关报道,主要集中在毛竹等温带散生竹中,筛选出PP2A、TIP41等作为毛竹不同组织基因表达定量分析的内参基因[9-11],但这些内参基因在不同发育时期的表达稳定性是未知的。针对巨龙竹不同秆形发育特点,本研究用qRT-PCR评价EF-1α、GAPDH、Actin、Tubulin、TIP-41、PP2A 6个内参基因在巨龙竹茎秆发育过程中的表达稳定性,筛选在巨龙竹不同生长阶段、不同秆形变异种群中最佳内参基因,为下一步解析参与巨龙竹秆形发育关键基因的表达模式提供技术基础。
HTML
-
以云南省西盟县中课种群(通直型)和孟连县景信种群(弯曲型)2个种群内01个月的笋(或幼竹)作为研究对象。巨龙竹发笋期(5月下旬至8月下旬)的定点观察结果显示,秆形变化主要出现在竹秆基部13 m内的竹节,1个月内秆形基本定型。因此,从每个类型种群中选择3个竹丛,每个竹丛选取出笋后5天、15天、30天的竹笋,分别代表秆形发育的初期、中期和后期。样品取自竹笋基部竹节,用干净的刀片切成小块立即放入液氮中,液氮处理后于-80℃贮存备用。
地点Site 茎秆类型Culm type 经度Longitude 纬度Latitude 海拔Altitude/m 采样时间Time 西盟县中课镇窝笼村 通直型 99°32′06″ 22°43′48″ 1 254 2 016-07-15 孟连县景信乡景信村 弯曲型 99°36′31″ 22°26′36″ 1 007 2 016-07-16 Table 1. Sampling information
-
RNA提取试剂盒(TransZol Up Plus RNA Kit),反转录试剂盒(TransScriptⅡ One-Step gDNA Removal and cDNA Synthesis SuperMix),qRT-PCR荧光染料(TransStart Top Green qPCR SuperMix)均购自北京全式金生物技术有限公司。
-
每个组织取3个不同的生物学重复。总RNA的提取参照TransZol Up Plus RNA Kit试剂盒说明书进行,提取的总RNA经2%琼脂糖凝胶电泳检测RNA完整性,Nanodrop 8000紫外分光检测仪检测OD值后直接用于反转录或保存于-80℃备用。
-
按照TransScript Ⅱ One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒说明书,各取样品的总RNA 7μL反转录成cDNA第一链。所得cDNA产物直接用于PCR和qRT-PCR或保存于-20℃备用。
-
使用Primer Premier 5.0软件设计候选内参基因的qRT-PCR引物,引物序列见表 2。引物由昆明硕擎生物技术有限公司合成,采用PAGE纯化。
基因
Gene name引物序列(5’→3’)
Primer sequence (5’→3’)解链温度
Tm/℃扩增产物长度Length of amplified product/bp PP2A F:CAGTCATCCCATGCTCTTGCT 60.6 158 R:GAAGCTGGCTTACGTGAGATGAA 59.8 EF-1α F:GCAACAAAACCACGCTTGAGA 59.6 186 R:GGGTCGTGTTGAGACTGGTG 59.5 GAPDH F:AGTCCACTGGTGTCTTCACTG 59.6 178 R:CAATTTGTGGTGCAGCTAGCATT 60.0 Actin F:TGCCTACATTGCCCTGGACTA 59.6 162 R:TCCATTCCAATGAAGGATGGCT 59.8 Tubulin F:GTCGATGCGCGAGAATACCT 59.4 186 R:GCAGTTGCAACCATCAAGACC 59.6 TIP-41 F:CAGCGAAATGGAAGTTCAGAAG 59.8 169 R:TGGTCATCAGTATCCTCCCAAC 59.7 注:正向引物(F)forwards primer,反向引物(R)reverse primer,解链温度(Tm)melting temperature。 Table 2. Primer sequences of six candidate reference genes for qRT-PCR analysis
-
利用表 2中的引物对6个候选内参基因进行普通PCR,体系为2×Taq PCR MasterMix 10 μL,混合cDNA 2.0 μL,正向和反向引物(浓度10 μmol·L-1)各1.0 μL,ddH2O补充体积至20 μL。反应温度程序为:95℃预变性2 min;94℃变性30 s,60℃退火30 s,72℃延伸30 s,45个循环;72℃延伸10 min。反应完成后用2%琼脂糖凝胶电泳检测各PCR产物。
-
qRT-PCR采用20 μL的反应体系:2×TransStart Top Green qPCR SuperMix 10 μL,各样品的cDNA 1.0 μL(阴性对照加ddH2O 1.0 μL),正向和反向引物(浓度10 μmol·L-1)各0.4 μL,用ddH2O补足体积至20 μL。反应程序为:94℃预变性30 s;94℃变性5 s,60℃退火30 s,40个循环;6595℃溶解曲线分析。实验设计3个技术重复。加样完成后迅速放入BioRad CFX 96 real-time PCR仪上进行反应。数据读取由仪器自动完成。
-
qRT-PCR扩增完成后,采用Ct值的方法[12]计算6个内参基因的表达量。利用内参基因评价软件GeNorm和NormFinder分别对不同基因进行稳定性评价,以确定理想的内参基因。
-
以筛选出的基因为内参,对巨龙竹木质素合成中PTAL(Phenylalanine/tyrosine ammonia-lyase)[13]基因的表达量进行定量分析,验证内参基因的有效性。体系和反应程序同1.2.5,实验设3个生物学重复,每个生物学重复设3个技术重复。根据项目组前期采用相同植物材料进行转录组测序获得的结果(待发表)和实时荧光定量分析之间基因表达量的相关性验证内参基因的有效性。
1.1. 材料
1.1.1. 供试材料
1.1.2. 主要试剂
1.2. 方法
1.2.1. 总RNA的提取
1.2.2. 反转录合成cDNA
1.2.3. 内参基因的引物设计
1.2.4. 候选内参基因的普通PCR
1.2.5. 候选内参基因的qRT-PCR分析
1.2.6. 数据分析及内参基因稳定性评价
1.2.7. 内参基因有效性验证
-
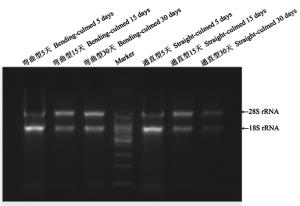
提取的总RNA经2%琼脂糖凝胶电泳结果显示如图 1,所提总RNA有28S、18S两条清晰的条带,条带无拖尾,RNA完整。经Nanodrop 8000紫外分光检测仪检测,A260/280值在1.72.0范围内,A260/230值大于2.0,RNA的纯度高,表明所制备的总RNA质量较好,满足试验需求。
-
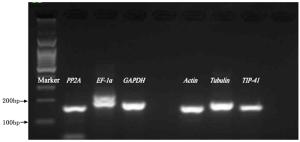
常规PCR结果显示,各内参基因均可见预期大小的目标条带(如图 2),且条带单一,无非特异性扩增,表明引物特异性良好,可用于后续qRT-PCR实验。
-
设置5个浓度梯度,3个技术重复,标准曲线相关系数R2均大于0.99,说明了线性关系好。扩增效率E在90%105%。熔解曲线有单一峰值,峰线窄而尖,说明引物特异性好,扩增产物单一。
-
对于不同样品或组织中基因表达的精确定量分析,选择合适的内参基因是关键,GeNorm软件和NormFinder软件是专业用来筛选内参基因的软件。
-
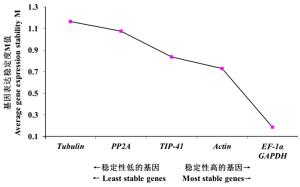
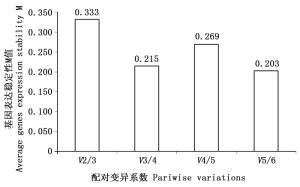
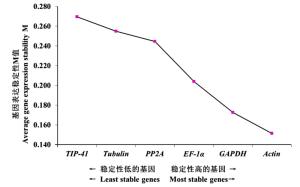
GeNorm软件程序采用几何平均算法,通过计算出每个内参基因稳定性的M值来筛选出稳定性较好的内参基因,M值越小内参基因的稳定性就越好,反之,稳定性越差。同时,通过归一化因子法计算出最适合的内参基因搭配组合[12, 14],来确定所需内参基因的最适数目,当Vn/(n+1)<1.5时,说明有n个基因可以作为qRT-PCR定量分析的内参基因,当Vn/(n+1)>1.5时,说明有n+1个基因可以作为qRT-PCR定量分析的内参基因。通过GeNorm分析,6个内参基因的稳定性由高到低的顺序为:EF-1α=GAPDH > Actin > TIP-41 > PP2A > Tubulin(图 3)。根据标准差数据分析(图 4),Vn/(n+1)均大于1.5,其中V5/6的M值最小,可以选择6个内参基因做组合。
-
NormFinder软件采用方差估计算法,先获得内参基因表达稳定值,再根据稳定值(M值)大小来筛选最适合的内参基因,M值最小的内参基因为最合适的内参基因[14-15]。通过NormFinder分析,6个内参基因的稳定性由高到低的顺序为:Actin > GAPDH > EF-1α > PP2A > Tubulin > TIP-41。
综上所述:通过使用GeNorm与NormFinder 2种软件对6个候选内参基因的稳定性评估结果得知GAPDH、Actin、EF-1α的稳定性最佳,可作为研究巨龙竹秆形发育过程基因表达分析使用的内参基因。
-
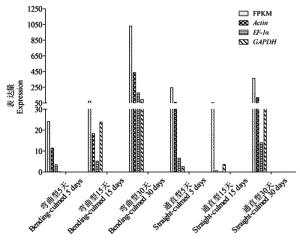
为了验证筛选的内参基因在巨龙竹茎秆发育过程中应用的有效性,选取木质素合成过程中的酶基因PTAL,分别以Actin、EF-1α和GAPDH为内参,对两种秆型巨龙竹不同发育时期的材料中的PTAL基因进行定量分析。结果如图 6所示,以Actin定量的PTAL基因表达量最高,GAPDH定量的PTAL基因表达量最低。根据前期转录组测序的结果,在弯曲型巨龙竹中,PTAL基因的FPKM值在发育5天、15天、30天的材料中分别为24.15、73.52和1 029.38;在通直型巨龙竹中分别为245.31、53.82和363.4。以不同内参基因获得的表达量与转录组测序获得的表达量进行相关性分析,相关性从大到小为Actin > EF-1α > GAPDH,相关系数分别为0.99,0.95和0.92,说明筛选出的3个内参基因有效性均较高。
2.1. 总RNA样品的质量分析
2.2. 内参基因的普通PCR分析
2.3. 标准曲线与熔解曲线的分析
2.4. 内参基因稳定性评价
2.4.1. GeNorm软件评价结果
2.4.2. NormFinder软件评价结果
2.5. 内参基因应用的验证
-
近年来,随着测序技术的快速突破,“基因组学”的研究越来越便捷,极大地促进了转录组等技术手段的迅猛发展,使之成为探索基因调控网络研究的得力工具。目前,荧光定量PCR(qRT-PCR)技术由于其更高的敏感性和特异性,已经成为基因表达深入研究的基本方法[6]。而合适稳定表达的内参基因的筛选将决定表达量数据的可靠性。本研究对不同变异类型、不同发育时期的巨龙竹竹笋样品进行内参基因筛选的结果对解析“通直型”和“弯曲型”巨龙竹差异发育的分子机理有重要意义。
在qRT-PCR反应中,合适内参基因的选择是决定目的基因精确定量分析的先决条件。理想的内参基因应在生物体不同的生长发育阶段、组织及各种外界因素条件下均恒定表达。然而,内参基因的适用性并不是普遍的,需要从多个内参基因中筛选出表达相对稳定的一个或一个以上作为研究对象的内参基因[15]。Zeng等[16]在对枸杞属两个种Lycium barbarum L.和L. ruthenicum Murray内参基因的筛选中,确定在不同的组织细胞中,表达最稳定的内参基因并不统一。对牡丹不同发育阶段种子和花瓣进行qRT-PCR内参基因筛选的结果也显示,在不同发育阶段及不同组织,最稳定的内参基因也不同[17]。而相同内参基因在禾本科的大麦、小麦和燕麦间的通用性研究结果表明,GAPDH、TUBB、18S rRNA在三者中都表达稳定,而EIF4A和TUBA分别在小麦和燕麦中表达稳定,说明并不是所有内参基因在亲缘关系相近的物种中都通用[18]。在竹亚科植物中,相关内参基因筛选的研究十分有限,而且并非所有的竹亚科植物都能通用一种内参基因。对毛竹成花基因PheTFL1表达分析的研究中,选择了PP2A作为毛竹PheTFL1基因定量分析的内参基因[9]。Fan[10]等从14个候选内参基因中选择TIP41和核苷酸通道结合蛋白基因(NTB)作为毛竹开花前后表达量分析的内参基因。
本研究中,选择EF-1α、GAPDH、Actin、Tubulin、TIP-41、PP2A作为巨龙竹的候选内参基因,其中EF-1α、GAPDH、Actin、Tubilin均为植物学研究中经典的内参基因。EF-1α主要参与快速增殖细胞的生长与增殖[19],GAPDH、Actin和Tubilin均与细胞骨架结构的形成有关[20-22]。PP2A和TIP-41是新挖掘的内参基因,PP2A主要参与细胞功能的调节[23],TIP-41主要参与细胞水分的转运[24]。这些内参基因已在多种植物研究中证明了其有效性[9-10, 18]。
-
通过候选内参基因的稳定性和有效性的验证分析与评估,在不同变异类型的巨龙竹笋期及其不同发育时期,表达稳定性最佳的内参基因是Actin、EF-1α、GAPDH。下一步将结合巨龙竹秆形发育转录组学的分析,利用筛选出的内参基因对后续筛选的目的基因进行定量分析,研究巨龙竹秆型发育过程中相关基因的表达模式。




 DownLoad:
DownLoad:




