-
木麻黄(Casuarina)作为我国最成功的外引树种之一,对防台风危害、沿海沙丘固定和贫瘠干旱的沿海沙地生态系统的恢复有不可代替的作用[1]。目前,华南沿海木麻黄防护林的种植材料主要是无性系,其中短枝木麻黄(Casuarina equisetifolia L. Johnson)无性系的栽种面积最大。近年营造的木麻黄防护林绝大部分使用主要几个木麻黄无性系,如福建省的“惠-1”、“平潭2号”等,广东省的“A-13”、“A-8”、“501”等,海南省的“宝-9”、“真-4”等[2],还有一些当地林场或私人苗圃场自己选育但未经遗传测定的无性系,因此,华南沿海木麻黄无性系可能存在数量不明、同物异名、命名混乱等现象。
传统的植物品种鉴定多采用形态学鉴定方法,但是植物的一些外观形态不稳定,易受到环境因素的影响,加上一些品种间的形态差异较小,采用形态学的鉴定方法具有时间长和可靠性低的缺点[3]。植物的DNA分子标记技术具有不受环境影响,且变异丰富等优点,被迅速应用到品种鉴定的实践中[4]。相较于其他分子标记,SSR标记是共显性标记,具有数量丰富、稳定性好、多态性高、基因组覆盖性好等优点,EST-SSR分子标记在保持了其优点的前提下,具有较强的种属间通用性,且因为它们来自于转录区域,因此更具保守性[5]。目前,在林业上,利用SSR分子标记进行品种鉴定的研究方法和技术已经比较成熟。如马尾松(Pinus massoniana.)[6]、柑橘(Citrus)[7]、刺槐(Robinia pseudoacacia)[8]、山杏(Armeniaca sibirica)[9]等利用SSR分子标记进行指纹图谱的构建,用于开展品种鉴定。在木麻黄树种上,Kullan等[10]研究开发了50对EST-SSR标记用于木麻黄遗传图谱的构建;许秀玉等[11]开发了235个EST-SSR标记,并利用其分析了木麻黄核心种质的遗传多样性与群体结构;胡盼[12]通过借助EST-SSR分子标记研究短枝木麻黄不同种源的遗传差异。目前,利用分子标记进行木麻黄无性系鉴定研究还比较少,基于EST-SSR分子标记进行无性系鉴定和构建木麻黄无性系指纹图谱的研究还未见报道。
本研究利用EST-SSR分子标记技术,对华南沿海主栽的短枝木麻黄无性系进行鉴定,摸清华南沿海正在使用的短枝木麻黄无性系及其数量,并构建基于EST-SSR分子标记技术的短枝木麻黄无性系指纹图谱,以期解决我国木麻黄无性系命名混乱等问题,为育成品种的精确鉴定提供技术支持,并对林业管理部门的科学决策、科研部门的种质资源引进与收集、木麻黄的遗传改良和新品种选育提供参考,对保护木麻黄的品种权和育种者的合法权益也具有重要意义。
HTML
-
本研究所用材料为华南三省(福建、广东、海南)沿海地区防护林主栽的短枝木麻黄无性系。沿着3个省沿海岸线的县、县级市或区,选择种植面积大的无性系人工林采样(图 1、表 1)。采集时间为2017年12月至2018年4月。选取生长健壮、无病虫害的嫩枝叶(木麻黄的叶片已退化成齿状,光合作用器官主要是针叶状的小枝条),放入硅胶干燥剂,带回实验室后保存于-80℃超低温冰箱,以备后续提取DNA。
采样地点
Provinces包含市、县或区
City, county, or district采集单株数
Sample quantity采样序号
Sampling number福建省(12个市、县或区) 连江县、长乐市、福清市、平潭县、莆田秀屿区、惠安县、漳浦县、云霄县、东山县、诏安县、龙海市和晋江市 31 131 广东省(16个市、县或区) 饶平县、南澳县、澄海区、惠来县、陆丰市、海丰县、惠东县、台山市、恩平市、阳东县、阳西县,电白县,吴川市、湛江坡头区和徐闻县 50 3281 海南省(14个市、县或区) 海口美兰区、文昌市,琼海市、陵水县、三亚崖州区、三亚海棠区、三亚天涯区、东方市、儋州市、临高县,乐东县、澄迈县、万宁市和昌江市 28 82109 Table 1. Sampling locations of C. equisetifolia clone
采集样品前,先向当地林业部门咨询当地所种植的木麻黄无性系的名称,咨询不到或者情况模糊的则只记录采样顺序号。采样过程中,注意采集各省份已育成并大规模推广应用的无性系作为鉴定的标准无性系。在福建省惠安赤湖林场采集到标准无性系“惠-1”和“平潭2号”,在福建省东山县赤山林场采集到“501”和“601”,在广东省吴川市采集了“宝-9”和“A-13”,在海南省岛东林场采集到“A-8”、“东-2”和“真-4”。最后共采集到109个木麻黄无性系单株和9个木麻黄标准无性系。
-
叶片材料的DNA提取采用改良后的CTAB法[13-14],提取的DNA稀释后-80℃保存备用。所用引物来源于课题组前期研发的21对引物以及Kullan等[10]开发的50对引物,共71对引物。选取8个短枝木麻黄无性系的DNA作为模板进行引物筛选,最终筛选出12对多态性高的引物(表 2)。
引物
PrimerSSR类型
SSR Type引物序列
Primer Sequence (5′-3′)预期片段大小
Expected product size/bp退火温度
Tm/℃C13 (CATCTT)3 F: ATGGGACATTTTGGTGAT
R: CTTTGCTTTAGGCGTTTT282 50 M03 (AG)11 F: GATCGTCAGGGGAAGAGTGA
R: CATCGGTTTCAGCGAGTACC290 60 M26 (AGA)15 F: GCCGTCTCTGAGAGTTTTGG
R: TAGAGACTGATCCTCGGCGT263 54 M27 (GAA)13 F: TTTTTAGATGGGTTTCCCCC
R: GAAATGAGCACTTCCTTGCC269 54 M32 (TC)12 F: AGCATGAAAGCAGGACCACT
R: AAGCCTGGCTGGCTCTGA179 60 M46 (TA)9 F: TGTTTCCATTTCTTATTTGCTTCA
R: TCAAATCTCCCAAACCCATC196 60 M36 (GAG)10 F: CGGAGGAGCTGTTGGTACTC
R: TACTTGCAGGTCGCTTTCCT248 60 M37 (GAA)16 F: TGCTTCAGCCTCACTCTTGA
R: CACCAACCCAATCAACTCAAC175 58 M38 (AG)20 F: TAACGGTCGACATGATTGGA
R: AAAATCATCGTCCAATCTCAGAA161 60 M39 (AG)18 F: CGTAAACGTGCGTGTGTTTT
R: TTGATTTTCGATTTCTCCGC160 60 M40 (AT)11 F: CAACATAATTGAATGCCCGA
R: GTCTGGCCCTGGATGTAAGA163 60 M41 (CT)17 F: CCTCCCCCTGTGGTTTTTAT
R: GAGATCTGAGGGTGCGAGAG170 58 Table 2. Information of primers for 12 SSR loci
-
参考Li等[15]降落PCR(Touchdown PCR)反应体系,采用在PCR反应体系中加入荧光dUTP的方法,利用12对EST-SSR引物对109个木麻黄无性系单株的DNA进行PCR扩增和毛细管电泳检测。降落PCR反应体系为10 μL,即:10 ng模板DNA、1 U TaqDNA聚合酶、1.21.6×buffer(Mg2+plus)、4~10 mmol·L-1dNTP、引物0.5~0.85 μmol·L-1、荧光dUTP 0.01 nmol·μL-1、ddH2O补平至10 μL。PCR反应程序如下:105℃热盖,94℃预变性5 min,95℃变性30 s,50~60℃退火30 s (每个循环降低0.5℃),72℃延伸30 s,循环20次,94℃变性30 s,60~70℃退火30 s,72℃延伸30 s,循环26次,72℃延伸10 min,4℃保存。PCR产物在ABI 3130x1遗传分析仪上进行分型。
-
原始数据采用Data Collection软件收集扩增片段信息。利用软件Data Former将数据转换成软件可识别格式,再利用POPGENE 1.31软件进行等位基因数(Na)、有效等位基因平均数(Ne)等的计算。利用EXCEL和遗传分析软件GenALEX6.5的Mutillocus功能对109个木麻黄无性系单株与9个标准无性系的基因分型结果进行比对,分析109个无性系单株中包含的无性系数量。
指纹图谱构建采用引物组合分析法[16]和指纹图谱代码方法[17],采用1~9字符进行赋值,删除鉴定性差(即多态性低)的等位基因,对12对引物扩增的所有等位基因进行筛选后,选择其中具代表性的基因位点进行赋值,保证每对引物下的编码只有一位数字,编码木麻黄指纹图谱代码。借助GenALEX6.5软件,通过不同引物的组合,达到以最少数量的引物完全区分所有待鉴定木麻黄无性系的目的,提高鉴定效率。通过软件NTSys2.10,根据Nei遗传距离对短枝木麻黄无性系进行UPGMA亲缘聚类。
1.1. 试验材料
1.2. 研究方法
1.2.1. 样品的DNA提取和引物筛选
1.2.2. 毛细管电泳和遗传分型
1.2.3. 数据统计分析与图谱构建
-
利用12对EST-SSR引物对109个木麻黄无性系单株进行扩增,共检测到50个等位基因,等位变异扩增片段长度范围为129~292 bp,每对引物可检测到的等位基因数为3~6个,平均等位基因数为4.2个;平均每对引物可检测到的有效等位基因数为2.96;每对引物可区分的无性系数量为2(M36)~5(M26)个,平均为3.4个,4个无性系在4个位点上具有特异等位基因(表 3)。
引物
Primer等位基因数Na 有效等位基因数Ne 多态性信息量PIC 区分材料数DV 扩增片段长度范围/bp C13 3 1.63 0.35 4 265277 M03 3 2.25 0.49 3 278292 M26 5 3.80 0.69 5 249267 M27 6 3.41 0.66 3 246270 M32 3 2.95 0.59 4 169179 M46 5 3.46 0.67 3 194210 M36 3 2.43 0.50 2 240258 M37 6 4.10 0.72 4 134167 M38 4 2.22 0.49 4 132154 M39 3 2.51 0.53 3 129139 M40 5 3.91 0.70 3 157169 M41 4 2.89 0.60 3 151179 平均值
Mean4.2 2.96 0.58 3.4 DV:区分材料数。DV: Distinguished varieties. Table 3. Genotyping of 109 C.equisetifolia clones using 12 pairs of EST-SSR primers
-
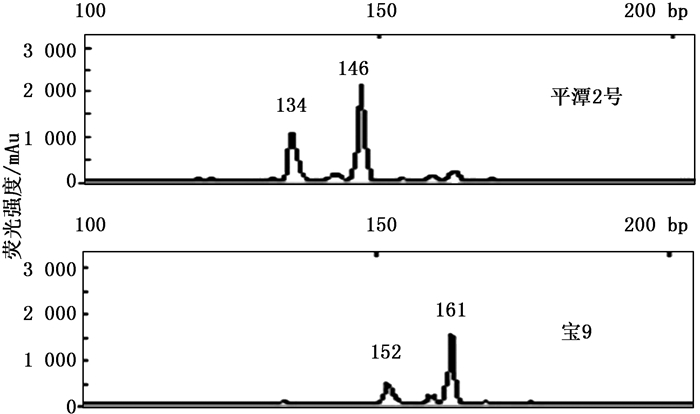
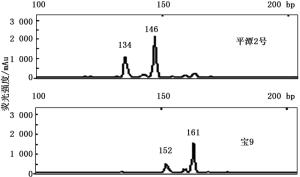
根据毛细管电泳的基因分型结果可知:12对引物可以将9个标准无性系(宝-9、A-8、501、东-2、短杂1号、A-13、601、平潭2号、惠-1)完全区分开来。以9个标准无性系的毛细管电泳结果(图 2)为参照,鉴定109个短枝木麻黄无性系单株。
在12个EST-SSR位点获得的基因型完全一致时,可以确定为同一个无性系(累积排除概率PE=0.998)。基因分型结果显示:109个木麻黄无性系单株最终归类为22个无性系(表 4)。根据分型结果,有13个不同地点采集的无性系单株跟“宝-9”标准无性系在12个位点的基因分型结果完全一致,可以被确定为“宝-9”无性系;另有16个单株被鉴定为“A-8”无性系,5个被鉴定为“501”无性系,4个被鉴定为“东-2”无性系,3个被鉴定为“短杂1号”无性系,3个被鉴定为“A-13”无性系,7个被鉴定为“601”无性系,11个被鉴定为“平潭2号”无性系,8个被鉴定为“惠-1”无性系。其中,被鉴定为“A-8”无性系的单株数量最多,在3个省的16个采样点都有大面积种植;还有39个无性系单株的基因型与9个标准无性系均不相同,被划分为13个不同的不知名无性系,暂且命名为UC-1(Unknown clone)~UC-13(表 4)。在采样过程中,未能从当地林业部门获得这13个无性系的来源地、育种者和命名等信息,可能是当地林业生产单位或私人苗圃自已选择优株进行无性繁殖获得的无性系。这些无性系通常未经过系统和严格的遗传测定便进行大面积造林应用,会对木麻黄沿海防护林造成巨大的潜在风险。林业管理部门应该采取措施对这种行为进行适当的指导和规范。
无性系名HH Clone name 基因分型相同的单株序号
Sample serial number with the same genotype单株数量
Sample number宝-9 13、39、82、85、87、91、94、95、97、98、100、104、108 13 A-8 3、7、8、10、11、22、27、61、70、81、89、96、99、101、103、107 16 501 4、38、68、69、71 5 东-2 24、46、47、83 4 短杂1号 18、42、43 3 A-13 45、50、52 3 601 30、36、40、41、44、48、84 7 平潭2号 2、17、19、20、23、25、28、29、31、72、106 11 惠-1 5、6、9、12、14、15、16、102 8 UC-1 21、62、86 3 UC-2 26、93 2 UC-3 33、88 2 UC-4 34、37、67 3 UC-5 35、65、105 3 UC-6 32、54、56、74 4 UC-7 49、75、109 3 UC-8 51、76、77 3 UC-9 55、57、58、73 4 UC-10 63、66 2 UC-11 1、59、60、64、78、80 6 UC-12 90、92 2 UC-13 53、79 2 Table 4. Identification of 109 C. equisetifoliac lones
结合采样情况,福建省采样31个,检测有11个无性系;广东省采样50个,检测有19个无性系;海南省采样28个,检测有12个无性系,结果显示:A-8、平潭2号、宝-9、东-2、601、UC-1等6个无性系在3个省间都互有苗木调用或引种种植;A-13、UC-4、UC-6、UC-8、UC-9等5个无性系只在广东省有种植,UC-12只在海南省有种植。可见,各无性系的分布范围各异、分布数量不均,其中,广东省使用的无性系较丰富,海南省和福建省所使用的无性系数量相对较少。参试木麻黄无性系的样品采集点覆盖了华南三省的42个市、县(区)中有大规模沿海防护林种植的区域,基本包括了所有大面积种植的木麻黄无性系。结果显示:局部地区存在无性系相对单一、多样性低等严重问题,说明应加强木麻黄新品种的选育工作。
-
采用引物组合分析法通过GenALEX6.5软件排序发现:只需7对引物就可达到将22个短枝木麻黄无性系完全区分的目的。依据这7对引物的毛细管电泳结果,构建了22个短枝木麻黄无性系的指纹图谱(表 5)。以平潭2号为例,代表的是平潭2号分别在引物M26的258 bp、M27的267和270 bp、M46的196和198 bp、M37的134和146 bp、M38的142 bp、M40的163和169 bp、M41的151和163 bp处检测到等位基因片段。对7对引物扩增的等位基因进行筛选后,选择了其中25个具代表性的等位基因进行赋值(表 6),编码了木麻黄无性系的指纹图谱代码(表 7),便于应用识别。编码后,每个无性系的DNA指纹图谱代码具有7位数字,分别代表该无性系在7对引物上扩增出的电泳峰值。如平潭2号,图谱代码为3321231,表示平潭2号在引物M26的258 bp(3)、M27的267 bp(3)、M46的196 bp(2)、M37的134 bp(1)、M38的142 bp(2)、M40的169 bp(3)、M41的151 bp(1)处的特异基因分型。
bp 无性系
Clone引物Primer M26 M27 M46 M37 M38 M40 M41 平潭2号 258/258 267/270 196/198 134/146 142/142 163/169 151/163 宝-9 249/267 267/270 194/196 152/161 132/142 157/161 163/179 A-8 252/267 267/270 196/198 134/146 142/154 163/169 151/163 东-2 258/258 261/264 196/198 152/161 142/142 163/167 151/163 短杂1号 267/267 267/270 194/196 152/161 132/142 163/169 159/163 601 258/258 267/270 196/198 134/146 132/142 163/169 151/163 惠-1 252/267 267/270 194/196 134/146 132/142 163/169 159/163 A-13 249/267 261/264 208/210 134/146 132/142 163/169 163/179 501 252/267 246/249 194/196 134/146 132/142 163/167 159/163 UC-1 249/267 267/270 196/198 134/146 142/154 163/169 151/163 UC-2 261/261 267/270 194/196 143/152 142/154 157/161 159/163 UC-3 261/261 267/270 194/196 152/161 132/142 163/167 159/163 UC-4 258/258 267/270 196/198 134/146 142/154 163/167 151/163 UC-5 249/267 267/270 194/196 143/167 142/154 157/161 163/179 UC-6 249/267 267/270 194/196 143/152 142/154 157/161 151/163 UC-7 252/267 246/249 208/210 134/146 132/142 163/169 163/179 UC-8 252/267 261/264 208/210 134/146 132/142 163/167 163/179 UC-9 249/267 267/270 194/196 143/152 142/154 157/161 159/163 UC-10 252/267 246/249 194/196 152/161 142/142 163/167 159/163 UC-11 252/267 261/264 208/210 134/146 142/142 163/169 151/163 UC-12 249/267 267/270 194/196 152/161 142/142 157/161 163/179 UC-13 252/267 246/249 194/196 143/152 142/146 163/167 151/163 Table 5. Fingerprints of 22 C. equisetifolia clones based on 7 primers
bp 引物Primer 编码Code 1 2 3 4 5 M26 249 252 258 261 267 M27 246 261 267 —— —— M46 194 196 208 —— —— M37 134 143 152 143 —— M38 132 142 146 154 —— M40 157 163 169 —— —— M41 151 159 163 —— —— Table 6. lleles selection and encoded standard
bp 无性系名
Clonal name指纹图谱代码
Fingerprint codes平潭2号 3321231 宝-9 1313113 A-8 2321431 东-2 3223221 短杂1号 5313132 601 3321131 惠-1 2311132 A-13 1231133 501 2111122 UC-1 1321431 UC-2 4312412 UC-3 4313122 UC-4 3321421 UC-5 1314413 UC-6 1312411 UC-7 2131133 UC-8 2231123 UC-9 1312412 UC-10 2113222 UC-11 2231231 UC-12 1313213 UC-13 2112321 Table 7. Fingerprint codes of 22 clones of C. equisetifolia
-
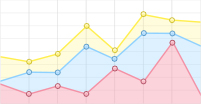
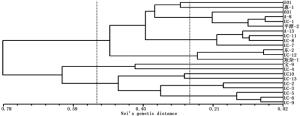
根据Nei遗传距离对22个短枝木麻黄无性系进行UPGAM聚类。由图 3可看出:22个无性系根据Nei遗传距离被分成了2个主要的组,大多数无性系两两之间的遗传距离小于0.3,说明这22个短枝木麻黄无性系相互间的亲缘关系较近,整体的遗传多样性较低,其中,部分的无性系间遗传距离极小,如A-8和UC-1、东-2和UC-12、UC-6和UC-9之间的Nei遗传距离不足0.10。
2.1. 华南沿海短枝木麻黄无性系的基因分型
2.2. 我国沿海主栽短枝木麻黄无性系的鉴别及其分布特点
2.3. 木麻黄的EST-SSR特征指纹图谱的构建
2.4. 22个短枝木麻黄无性系亲缘关系分析
-
根据建立SSR标记的序列性质不同,SSR标记可分为基因组SSR(gSSR)和表达序列标签SSR(EST-SSR)[18]。EST-SSR标记具有gSSR标记的共显性、重复性高、多态性高等优点[19-20],且较之gSSR标记EST-SSR具有较强的种属间的通用性[5]。近年来,许多物种的大规模cDNA测序并在网上公开释放,大大增加了EST-SSR标记的开发能力[21]。在GenBank等数据库可以获得大量相关植物的表达序列标签且数据库仍在不断更新中,为其EST-SSR引物的开发提供了极大便利。因此,EST-SSR逐渐成为植物遗传多样性分析、系谱分析、品种鉴定及DNA指纹图谱构建中广泛应用的分子标记。
稳定性高、多态性好的引物可以得到更好的鉴定效果[22]。本文的12对EST-SSR引物在木麻黄的109个木麻黄无性系单株中总共扩增出50个等位基因,平均每对为4.2个,这与许秀玉等[11]利用226对EST-SSR标记在短枝木麻黄中扩增得到的5.4个平均等位基因数相近,低于胡盼[12]利用13个EST-SSR标记在29个短枝木麻黄种源中扩增得到的23.69个平均等位基因数和Kullan等[10]在短枝木麻黄与山地木麻黄扩增得到的17个平均等位基因。在12个位点中,PIC值(多态性信息量)变幅为0.35~0.72,根据引物多态性信息量的评价标准(高:PIC>0.5;中:0.5>PIC>0.25;低:PIC<0.25)[23],这12对引物中有9对(75%)的PIC高于0.5,剩余3对的PIC也高于0.25,表明其均有较高的多态性,PIC值越大,引物鉴定能力越强[24],说明所选EST-SSR引物能够较好地用于短枝木麻黄无性系指纹图谱的构建。本研究12对EST-SSR引物扩增出的平均等位基因数稍低可能是由于参试无性系较少(实际是22个无性系)引起的,对此有待进一步研究。
-
毛秀红等[8]对刺槐的研究,前后2次各利用9对不同的引物均无法区分“鲁刺62”、“鲁刺90”、“鲁刺57”、“鲁刺103”这4份无性系,由此推断它们可能是同物异名。本文研究发现,很多具有不同名字的短枝木麻黄无性系其实是属于同一个无性系(其12个扩增位点的等位基因完全相同),如广东选育的“601”(采样序号36)无性系和海南的“真-4”(采样序号84)无性系是同一个无性系(注:20世纪80年代初601无性系选育时,海南岛属于广东省),海南选育的“东-2”(采样序号83)和广东的“BF-3”(采样序号47)为同一个无性系,还有一些具有当地名字的无性系实际是从其他省份或地区引进后被重新命名,存在重复命名的现象。曾乐[25]对大麦进行品种鉴定时发现,不同的大麦品种由于遗传背景接近导致其在外观形态特征上很相似,相比DUS测试鉴定法,SSR分子标记鉴定法具有快速、方便、不受环境条件影响等优点。短枝木麻黄无性系多种植在海边,生长环境较恶劣,形态性状极易受环境影响。本文实践证明,利用EST-SSR标记技术开展木麻黄无性系的鉴别,可以精确鉴定外观形态上近似的无性系,解决命名混乱的现象,鉴定效率更高,鉴定结果更可靠,有利于木麻黄无性系选育和鉴定工作的开展。
-
DNA指纹图谱是快速鉴别植物品种时的有效工具,效率较DUS鉴定法更加准确快速,错误率更低[26-27],能在各类植物中普遍应用。DNA指纹分析方法一般有特征谱带法、引物组合法和核心引物组合法[16],其中,特征谱带法仅需用1对引物就可以鉴别品种,简单方便,实用价值大;但是,并不是所有的品种都有特征谱带,本研究只有4个无性系在4个位点上具有特异等位基因,而金玲等[9]通过155对引物的扩增,于97个山杏无性系中发现89个无性系的特异等位基因。若想获取其他无性系的特征谱带,还需要进行大量的引物筛选工作;而引物组合法通过不同引物的组合,利用相对少量的引物就可以区分全部材料,避免了大量引物筛选的繁重工作量。本研究中,利用7对引物可将22个木麻黄无性系完全区分开,引物组合的鉴定效率高于刘欢等[28]利用6对EST-SSR引物组合完全区分10个多花黑麦草材料,而低于金玲等[9]利用5对SSR引物可全部区分97个山杏无性系和赵阿风[6]利用8对SSR引物组合可全部区分120个马尾松无性系的鉴定效率,这可能与鉴定材料之间的亲缘关系远近程度有关。本文所鉴定的无性系之间亲缘关系较近,鉴定难度大,所需引物数量也较多。如毛秀红等[8]与袁存权等[29]同样利用8对多态性高的引物分别对来自不同地市的49份刺槐材料和来自同一林场的48份刺槐材料进行鉴定,前者所得等位基因数是后者的2.04倍,分析原因可能与后者所用材料之间的亲缘关系相近有关,对此应该做进一步研究。
3.1. EST-SSR标记的优点和效果
3.2. 品种鉴定
3.3. 基于EST-SSR分子标记的DNA指纹图谱构建
-
本文开展了基于EST-SSR标记的我国华南沿海地区短枝木麻黄无性系的鉴定及指纹图谱构建的研究,筛选出12对多态性较高的EST-SSR引物;109个短枝木麻黄无性系单株被鉴定为分别属于22个无性系;各无性系在3个省间互有引种,引种过程中存在重复命名的情况,各无性系的分布范围各异、分布数量不均,海南和福建两省存在栽培无性系数量较少的情况;采用7对引物扩增谱带的组合构建了22个短枝木麻黄无性系的DNA指纹图谱,建立了基于EST-SSR标记的短枝木麻黄无性系指纹鉴定体系;目前所种植的短枝木麻黄无性系之间整体亲缘关系较近,遗传多样性不高,对此应加强新品种的选育。



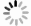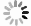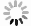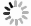
 DownLoad:
DownLoad: